Les 1
Leeruitkomsten
- De student kan informatie over een gen opzoeken in de NCBI gene database.
- De student kan informatie over de sequentie van transcripten opzoeken in de NCBI nucleotide database.
- De student kan (delen van) sequenties van transcripten verkrijgen via de NCBI nucleotide database.
Voorbereiding
Biologische achtergrondinformatie
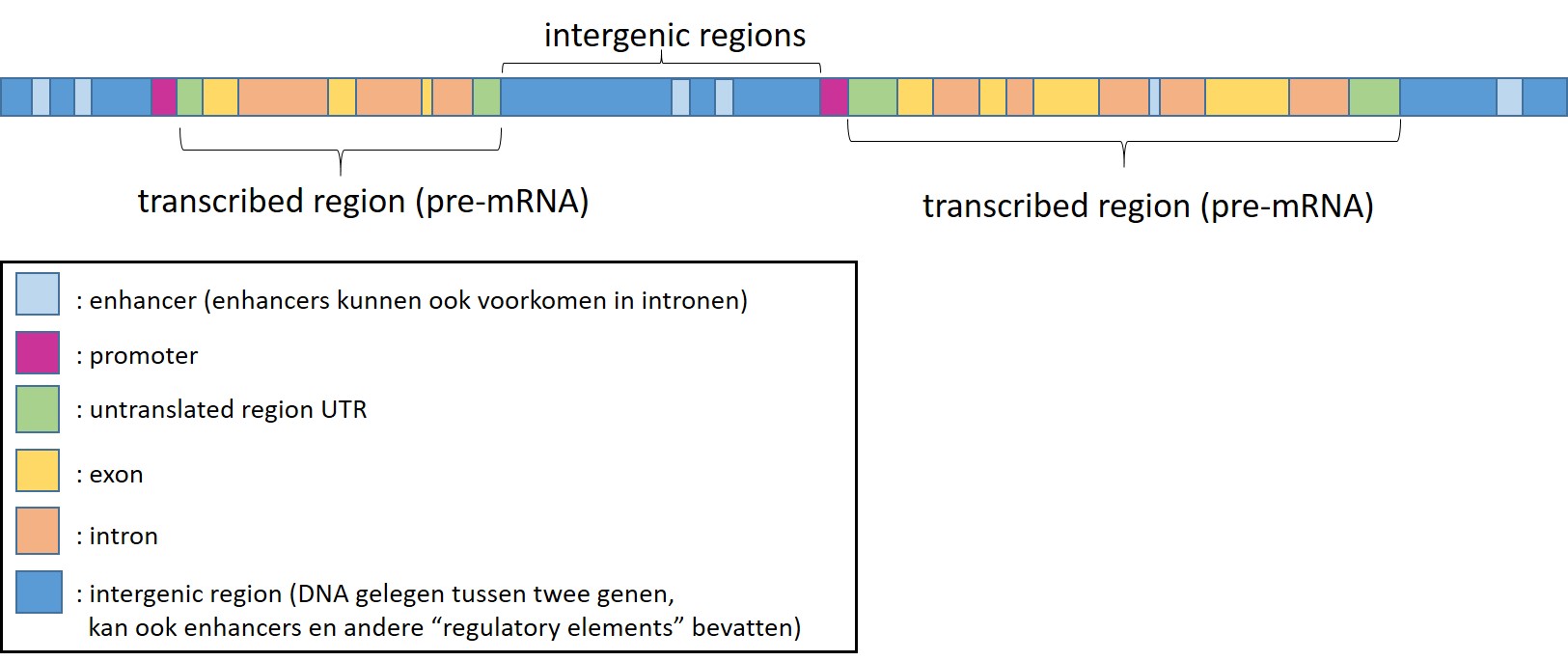
Het menselijk genoom
Genetische informatie is opgeslagen in het DNA van de cel. Bij de mens is dit DNA verpakt in 23 paar chromosomen. De complete DNA sequentie van al dat DNA noemen we het genoom. Het genoom bevat verschillende elementen (zie het onderstaande figuur). Het genoom bestaat voor 2% uit eiwit-coderende genen (die op hun beurt weer bestaan uit exonen, intronen en UTRs). De overige 98% van het DNA (intergenic regions) werd vroeger omschreven als ‘junk DNA’, omdat er gedacht werd dat het niets deed. Nu weten we dat er zich in dit DNA veel regulatoire elementen bevinden die bepalen in welke mate genen tot expressie komen. Bovendien worden delen van dit DNA ook afgeschreven tot RNA dat niet codeert voor eiwitten.
Splicing
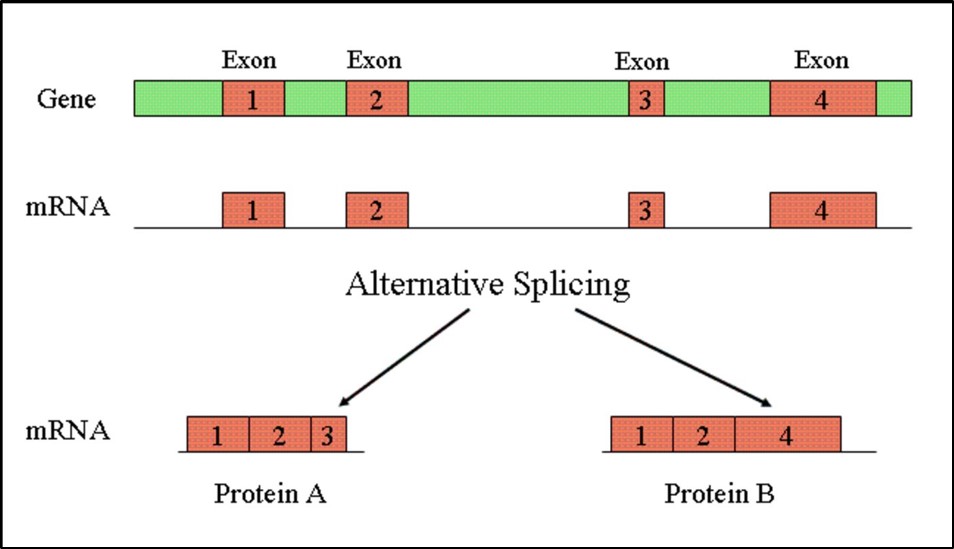
Eiwit-coderende genen bestaan uit intronen en exonen. Tijdens het splicing proces worden de intronen uit het premature RNA molecuul verwijderd en worden de exonen verbonden tot een transcript. Dit transcript kan vervolgens worden afgelezen door de ribosomen om een eiwit te vormen.
Eén gen kan meerdere transcripten vormen via alternative splicing. Dit betekent dat de verschillende transcripten van een gen verschillende combinaties van exonen bevatten (zie het onderstaande figuur). Op deze manier kan de DNA sequentie van één gen worden gebruikt om verschillende eiwitten te maken.
De structuur van het messenger RNA
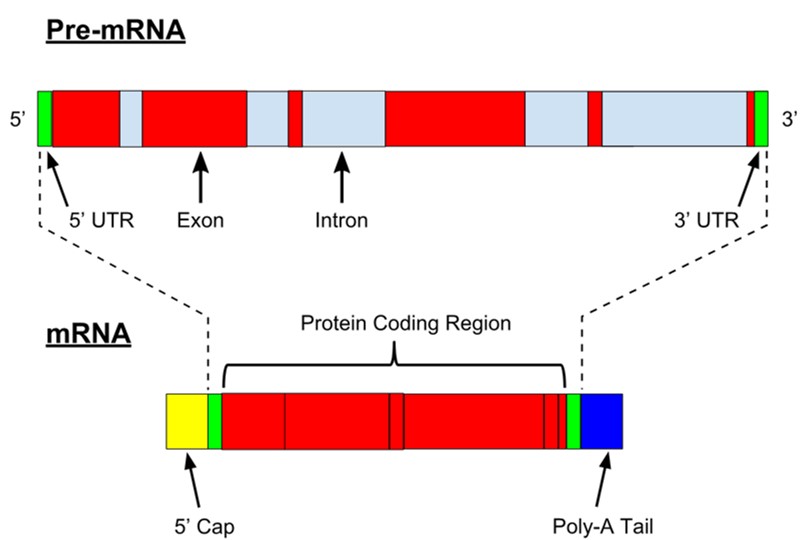
Via splicing worden de intronen uit het pre-messenger RNA verwijderd en blijven enkel de exonen over (zie het figuur hieronder). Deze exonen vormen samen het messenger RNA (mRNA). Voordat het mRNA naar de ribosomen gaat worden er ook nog een 5’ cap en een poly-A staart aan vastgemaakt om het mRNA molecuul stabieler te maken.
De exonen vormen dus samen de nucleotide sequentie van het mRNA. Een deel van deze sequentie zal worden worden afgelezen door de ribosomen om het eiwit te maken. Dit deel de sequentie, de coderende sequentie (CDS), begint met het startcodon (ATG) en eindigt met een stopcodon (TAA, TAG of TGA). Het eerste en laatste deel van de mRNA sequentie codeert niet voor het eiwit; deze delen vormen de 3’ untranslated region (UTR) en de 5’ UTR.
De NCBI database
Eén van de grootste databases voor biologische data is de NCBI database. De NCBI database bestaat uit verschillende kleinere databases. Twee voorbeelden hiervan zijn de gene en nucleotide databases die we in deze les gaan bekijken. Al deze kleinere databases zijn binnen NCBI met elkaar verbonden en er zijn vaak ook verwijzingen naar andere databases buiten NCBI aanwezig. De NCBI database is daarmee een uitstekend startpunt voor het zoeken naar biologische informatie.
De NCBI gene database
Als we informatie willen zoeken over een specifiek gen, dan is de NCBI gene database een goed startpunt. Met deze database kunnen we (onder andere) de volgende vragen beantwoorden:
- Waar ligt het gen in het genoom (welk chromosoom en welke positie)?
- Wat is de grootte van het gen?
- In welke weefsels komt het gen tot expressie en in welke mate komt het tot expressie?
- Welke genen zijn homologen van het gen?
- Wat is de functie van het gen?
Hieronder zullen we kijken naar deze vragen en zien waar in de NCBI gene database het antwoord op deze vragen te vinden is. Als voorbeeld zullen we in de uitleg hieronder het gen HK1 (hexokinase) bekijken. In de opdrachten ga je zelf aan de slag met het gen SMAD2.
Een gen opzoeken in de NCBI gen database
In de onderstaande video wordt uitgelegd hoe je een gen kunt opzoeken in de NCBI gene database. In de video wordt getoond hoe je het gen kunt opzoeken met het gensymbool. Je kunt in plaats van het gensymbool ook de gennaam of andere omschrijvingen gebruiken.
Algemene informatie over het gen
In de onderstaande video wordt uitgelegd waar je algemene informatie over het gen kunt vinden in de NCBI gene database.
Elk gen heeft in de NCBI database een unieke gene identifier. Deze identifiers zijn handig omdat de gennaam of het gensymbool hetzelfde kunnen zijn in verschillende soorten, maar de gene identifier is altijd uniek voor één gen.
Opdracht 1
Wat is de gen identifier van het SMAD2 gen?
Klik hier voor het antwoord
De identifier is te vinden bovenaan de pagina van het gen:
Opdracht 1
Lees de samenvatting van het SMAD2 gen. In welk moleculair proces speelt dit gen een rol?
Klik hier voor het antwoord

De genoompositie en verschillende transcripten
In de onderstaande video wordt uitgelegd waar je de genoomlocatie van het gen kunt vinden op de NCBI gene pagina. Ook wordt uitgelegd hoe je informatie kunt vinden over de verschillende transcripten van het gen.
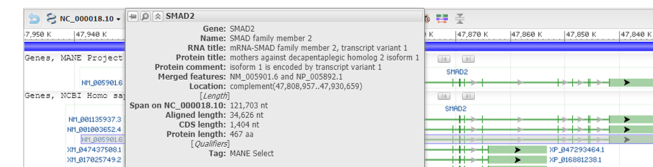
Alle transcripten en eiwitisoformen in de NCBI database hebben hun eigen, unieke NCBI identifier. De identifier van transcripten begint met NM_ (waarbij de ‘M’ staat voor mRNA). De identifier van eiwitten begint met NP_ (waarbij de ‘P’ staat voor protein). Als je meer informatie wilt over een bepaald transcript of eiwitisoform, dan moet je de identifier gebruiken om die informatie te vinden.
Opdracht 1
Hoeveel aminozuren bevat het product van SMAD2 transcript NM_005901.6?
Klik hier voor het antwoord
Het eiwit bestaat uit 467 aminozuren:
De expressie in verschillende weefsels
Op de NCBI gene pagina vind je ook informatie over de expressie van het gen in verschillende weefsels. Deze gegevens zijn gebaseerd op RNA sequencing datasets. Met RNA sequencing kun je voor elk gen bepalen hoeveel RNA er aanwezig is in een weefsel. Om de data voor verschillende weefsels/experimenten met elkaar te kunnen vergelijken, is de data genormaliseerd en zijn de waarden weergegeven als RPKM waarden. Hoe hoger de RPKM waarde, hoe hoger de expressie. De aanname is dat hoe hoger de expressie is (dus hoe meer transcripten er voor een gen aanwezig zijn), hoe meer eiwit er wordt gemaakt en hoe relevanter het gen is voor het weefsel.
In de onderstaande video wordt getoond hoe je de expressiedata kunt raadplegen op de NCBI gene pagina.
Opdracht 1
In welk weefsel komt SMAD2 het meest tot expressie? Wat is de gemiddelde expressie (in RPKM) in dit weefsel?
Klik hier voor het antwoord
Het eiwit komt het meest tot expressie in de schildklier (thyroid). De gemiddelde expressie is ongeveer 6.5 RPKM.De orthologen van een gen
Het gen hexokinase komt ook in andere organismen voor. Equivalenten van een menselijke gen in andere organismen noemen we orthologen. Orthologen zijn evolutionair verwant aan elkaar. De functie van de orthologen in de verschillende organismen is vaak hetzelfde, omdat de functie van orthologen evolutionair geconserveerd is.
In de onderstaande video wordt uitgelegd hoe je de orthologen van een gen kunt vinden via de NCBI gene pagina.
Opdracht 1
Wat is de gene identifier voor de ortholoog van SMAD2 in de rat?
Klik hier voor het antwoord
De gene identifier voor de ortholoog in de rat is 29357.De functie van een gen
Voor de meeste genen van de mens geldt dat er veel informatie beschikbaar is voor het eiwit waar het gen voor codeert. Zo weten we vaak de functie van het eiwit, we weten bij welke processen het eiwit betrokken is en we weten waar in de cel het eiwit zijn functie uitoefent. Om al deze informatie te structureren, zijn er gene ontology (GO) termen gedefinieerd. Deze termen geven informatie over de moleculaire functie, de locatie in de cel en de biologische processen van een gen en het bijbehorende eiwit. Deze GO termen zijn overkoepelende termen en kunnen gebruikt worden voor meer dan één gen en worden ook gebruikt voor andere soorten organismen dan de mens. In de onderstaande video wordt uitgelegd waar je de GO termen kunt vinden op de NCBI gen pagina:
Opdracht 1
Voor SMAD2 is bij het onderdeel component van de gene ontology sectie aangegeven dat het eiwit zowel in het cytosol als in de nucleus aanwezig is. Kun je dit verklaren?
Klik hier voor het antwoord
SMAD2 is een transcriptiefactor en beweegt na het ontvangen van een signaal van het cytosol naar de nucleus om daar de genexpressie te beïnvloeden.De NCBI nucleotide database
Voor sommige toepassingen in het biologische onderzoek (bijvoorbeeld het ontwerpen van PCR primers) hebben we de nucleotide sequentie van een gen of transcript nodig. De nucleotide sequenties voor alle menselijke genen zijn beschikbaar in de NCBI nucleotide database. We zullen nu bekijken hoe je deze informatie kunt opzoeken en gebruiken.
Transcripten opzoeken in de database
Vaak zijn we geïnteresseerd in de sequentie van transcripten. In de onderstaande video wordt uitgelegd hoe je de sequentie voor een bepaald transcript kunt opzoeken:
Transcriptinformatie en -sequenties opzoeken in de database
Op de pagina voor een transcript kun je veel informatie vinden over de sequenctie. In de onderstaande video wordt getoond welke informatie waar te vinden is. Verder wordt getoond hoe je de sequentie van (delen van) het transcript kunt downloaden.
Een algemeen bestandstype om nucleotide of aminozuur sequenties op te slaan is het FASTA bestand. Een FASTA bestand is een tekstbestand waarvan de eerste regel begint met een > teken, gevolgd door informatie over de sequentie (bijvoorbeeld de identifier, de naam van het gen en de genoomcoördinaten). Op de tweede regel begint de sequentie.
Opdracht 1
Zoek het SMAD2 transcript op met identifier NM_005901.6. Beantwoord voor dit transcript de volgende vragen:
- Wat zijn de coördinaten van het CDS van dit transcript?
- Wat zijn de coördinaten van het eerste en laatste exon van dit transcript?
- Wat valt je op als je de coördinaten van het eerste en laatste exon vergelijkt met de coördinaten van het CDS?
Klik hier voor het antwoord
- Coördinaten van het CDS: 353-1756.
- Coördinaten van eerste exon: 1-299.
- Coördinaten van laatste exon: 1633-34626.
- Het eerste exon ligt helemaal voor het CDS. Het laatste exon overlapt deels met het einde van het CDS, maar een groot deel ligt voorbij het CDS. Het eerste exon is onderdeel van de 5’ UTR, en het laatste exon vormt het laatste deel van het CDS en het 3’ UTR.
Opdracht 1
Download for the transcript NM_005901.6 (see previous exercise) the CDS sequence as a FASTA file.
Hint: om de sequentie te downloaden kun je de FASTA opvragen via de NCBI nucleotide database en dan kopiëren naar Notepad (Windows) of textedit (Mac). Vandaaruit kun je het tekstbestand opslaan op je laptop en bewaren voor een volgende les.
Werkcollege
Casus Les 1
In deze casus gaan we kijken naar het gen ACSL1 in de mens. Beantwoord voor dit gen de volgende vragen:
Algemene informatie opzoeken over het gen
- Wat is de gen identifier van ACSL1?
- Op welk chromosoom is ACSL1 aanwezig?
- Hoeveel exonen heeft ACSL1?
- In welke weefsels komt ACSL1 (vooral) tot expressie?
- Wat is de functie van het eiwit waar ACSL1 voor codeert? En waar in de cel is het eiwit actief?
- Wat is de gen identifier van de ACSL1 ortholoog in het konijn (Oryctolagus cuniculus)?
Informatie over de transcripten
- Wat is de lengte van transcript 2 (NM_001286708.2)? En wat is de lengte van transcript 18 (NM_001381889.1)?
- Wat is de lengte van de coderende sequentie (CDS) van transcript 2 en transcript 18?
- Als het goed is is de gevonden CDS lengte voor zowel transcript 2 als 18 korter dan de gevonden transcriptlengte. Kun je verklaren waarom dit zo is?
Sequenties voor de transcripten downloaden
- Download de sequenties voor de coderende sequentie van transcript 2 en transcript 18. Zorg ervoor dat je de sequenties opslaat in FASTA formaat.