Les 4
Leeruitkomsten
- De student kan qPCR primers ontwerpen met Primer-BLAST voor het bepalen van de genexpressie met qPCR.
- De student kan de specificiteit van de ontworpen primers controleren met de UCSC in silico PCR tool.
Voorbereiding
Biologische achtergrondinformatie
Celtypen verschillen in de expressie van genen
In ons lichaam zijn er honderden verschillende celtypen. Al deze cellen hebben hetzelfde genoom, d.w.z. het DNA in de celkern van de cellen is voor alle celtypen hetzelfde. Toch verschillen de celtypen van elkaar in fenotype. Dit komt door de verschillen in de genexpressie.
Genexpressieverschillen ontstaan door verschillen in de transcriptie van genen. Tijdens de transcriptie wordt het DNA van een gen overgeschreven naar RNA. De RNA moleculen verlaten de celkern en worden tijdens de translatie door de ribosomen in het cytoplasma gebruikt om eiwitten te maken. Cellen kunnen de transcriptie van genen uit- of aanzetten, en als de transcriptie aan staat kunnen cellen ook de transcriptie ‘harder’ of ‘zachter’ zetten. Deze transcriptieverschillen werken door in de eiwitten die een cel heeft: als de transcriptie van een gen hoger is, betekent dat over het algemeen dat een cel meer van het bijbehorende eiwit maakt. Op deze manier bepalen de transcriptieverschillen via de eiwitten de verschillen in fenotype, want het zijn de eiwitten die het fenotype veroorzaken.
Genexpressie meten met qPCR
We kunnen de expressie van genen in een cel op verschillende manieren meten. Een van die manieren is Reverse Transcription quantitative real-time PCR (RT-qPCR of qPCR). Met deze methode kun je de expressie van specifieke genen in de cel meten. Een qPCR experiment bestaat uit de volgende stappen:
- RNA isolatie. Het RNA wordt geïsoleerd uit de cellen waarin we geïnteresseerd zijn.
- cDNA synthese. Het RNA wordt omgezet naar cDNA. De PCR reactie van de qPCR kan namelijk alleen op DNA worden uitgevoerd.
- PCR en het gelijktijdig meten van productvorming. Het cDNA wordt gemengd met primers die specifiek zijn voor het gen/transcript waarin we geïnteresseerd zijn, een DNA polymerase en een intercalerende fluorescente stof (bijv. SYBR-green). Vervolgens voert de qPCR machine de PCR uit op dit mengsel. Tijdens elke PCR cyclus wordt het cDNA waaraan de primers binden, verdubbeld. De intercalerende fluorescente stof gaat tussen de strengen van het PCR product zitten en levert zo een fluorescent signaal op. Aan het eind van elke PCR cyclus wordt dit signaal door de qPCR machine gemeten.
Met qPCR kun je de vorming van het PCR product over de tijd volgen. De primers zorgen ervoor dat alleen specifieke cDNA moleculen worden geamplificeerd (in deze les zul je leren hoe je deze primers kunt ontwerpen). Als er in het begin van de PCR meer van dat specifieke cDNA aanwezig is, zal het fluorescente signaal sneller zichtbaar worden dan wanneer er weinig van dat cDNA aanwezig is. Hoe sneller je dus fluorescentie meet, hoe meer cDNA er in het monster aanwezig was.
Referentiegenen in een qPCR experiment
Zoals bij alle experimenten is het ook bij een qPCR experiment dat je controles meeneemt. De controles voor een qPCR experiment zijn als volgt:
- Een negatieve controle. Dit is een sample met water in plaats van cDNA. Hierin verwacht je geen signaal. Als er wel een signaal is, dan duidt dat op vervuiling van je reagentia met cDNA.
- Een positieve controle. Dit is een sample met daarin primers en cDNA waarvan je weet dat het een signaal geeft (omdat je dit in een eerder experiment hebt gemeten). Als je dit signaal niet meet, weet je dat er iets niet goed is gegaan met het inzetten van de PCR of dat een van de reagentia niet werkt.
- Een loading controle. Dit is een sample waarbij je het cDNA mengt met primers gericht tegen een referentiegen waarvan de expressie stabiel is in de verschillende condities.
Het belang van de loading controle/referentiegenen wordt in de onderstaande video uitgelegd:
Een paar opmerkingen bij de video:
- Referentiegenen worden ook wel huishoudgenen genoemd, omdat deze genen vaak een algemene functie hebben in de cel (dus bij ‘het huishouden’ van de cel horen).
- Het is belangrijk dat je het referentiegen kiest dat past bij jouw experiment. Het referentiegen moet zo gekozen worden dat het in alle te meten condities dezelfde expressie heeft. Er zijn géén universele referentiegenen die je altijd kunt gebruiken! Je kunt literatuur gebruiken om een goed referentiegen te selecteren. Vaak is het noodzakelijk om experimenteel te testen of het referentiegen ook daadwerkelijk bruikbaar is in jouw experiment.
- Je gebruikt referentiegenen om onderscheid te kunnen maken tussen echte genexpressieverschillen en verschillen die ontstaan door de voorbereidende stappen in het qPCR experiment (RNA isolatie en cDNA synthese). Het blijft echter heel erg belangrijk om verschillen door technische fouten zoveel mogelijk te voorkomen, bijvoorbeeld door het RNA van alle monsters tegelijkertijd te isoleren en door de cDNA synthese van alle monsters op hetzelfde moment uit te voeren.
Ontwerpeisen voor qPCR primers
In principe gelden voor qPCR primers dezelfde ontwerpeisen als voor PCR primers (zie de vorige les). Een aanvullende ontwerpeis is dat de primers op verschillende exonen moeten binden. Het gaat hier om twee opeenvolgende exonen: als bijvoorbeeld de forward primer op exon 1 bindt, dan moet de reverse primer op exon 2 binden (zie het onderstaande figuur). Dit is belangrijk om onderscheid te kunnen maken tussen PCR producten afkomstig van cDNA (het DNA dat we hebben gemaakt uit het mRNA en waarin we dus geïnteresseerd zijn) en vervuiling van genomisch DNA. Als de primers binden aan genomisch DNA, zorgt het tussengelegen intron ervoor dat het product veel groter wordt. Dit product wordt tijdens de qPCR niet gemaakt, omdat de amplificatietijd bij een qPCR cyclus niet zo lang is. Op die manier ontstaat er dus alleen een product als de primers binden aan het cDNA en wordt ons qPCR signaal dus niet verstoord door de eventuele aanwezigheid van genomisch DNA.
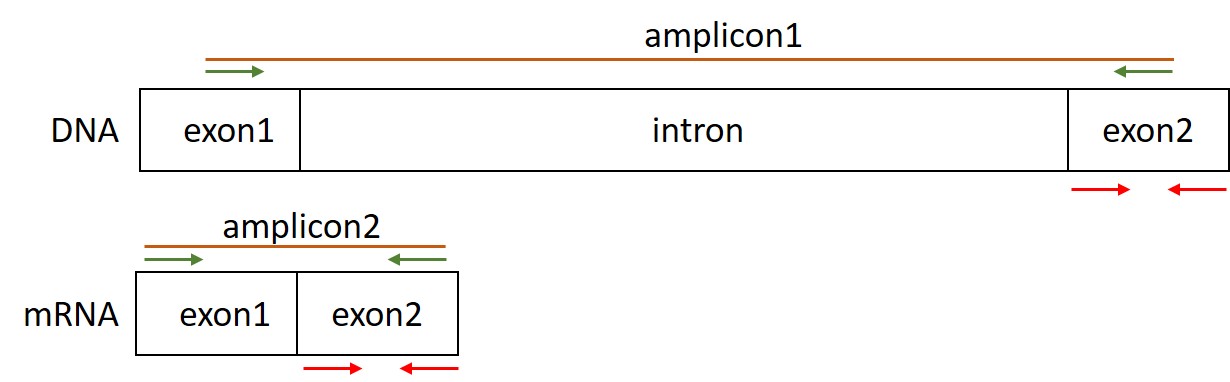
Exonen selecteren voor qPCR primers
Om primers te ontwerpen voor qPCR, moeten we eerst voor een gen twee opeenvolgende exonen selecteren. Dit kan een uitdaging zijn als er meerdere transcripten zijn voor een gen. In dat geval willen we namelijk twee exonen kiezen die in zoveel mogelijk transcripten voorkomen, zodat we de genexpressie kunnen bepalen onafhankelijk van het transcript dat aanwezig is in de cel.
In de onderstaande video wordt uitgelegd hoe je dit kunt doen a.d.h.v. het MAP2 gen, een gen dat specifiek tot expressie komt in neuronen:
Voor het ontwerp van de qPCR primers voor MAP2 kunnen we dus aan de slag met exon 6 en 7 van transcript NM_001375493.1.
Opdracht 4
We willen voor de neuronen die we in kweek hebben niet alleen kijken naar de expressie van MAP2 (een algemene neuronale marker), maar ook naar de expressie van VGLUT1 (een marker specifiek voor glutamaterge neuronen). Bepaal voor dit gen welke exonen gebruikt kunnen worden voor het ontwerpen van qPCR primers en zoek de bijbehorende coördinaten op.
Klik hier voor het antwoord
Het officiële gensymbool voor dit gen is SLC17A7. Op de NCBI genpagina van dit gen is te zien dat er voor dit gen maar één transcript bekend is. Voor dit transcript, NM_020309.4, kiezen we hier voor exon 2 en exon 3 (maar andere combinaties van opeenvolgende exonen zijn ook bruikbaar). De coördinaten van deze exonen zijn:
- Exon 2: 211 t/m 463.
- Exon 3: 464 t/m 582.
Opdracht 4
Om de qPCR uit te voeren voor VGLUT1 is een referentiegen nodig. Een veelgebruikt referentiegen is GAPDH. Bepaal voor dit gen welke exonen gebruikt kunnen worden voor het ontwerpen van qPCR primers en zoek de bijbehorende coördinaten op.
Klik hier voor het antwoord
Er zijn voor dit gen meerdere exoncombinaties die in alle transcripten voorkomen. De exonen die hier gekozen zijn zijn gearceerd in het onderstaande figuur:
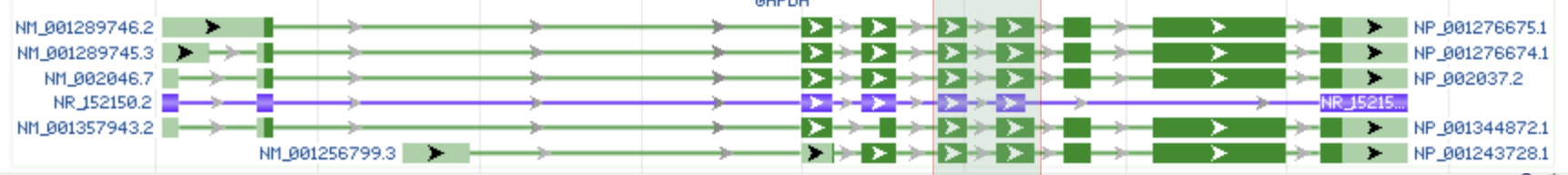
We nemen het eerste transcript, NM_001289746.2, als template voor het primerontwerp. In dit transcript lijkt het te gaan om exon 4 en 5. Om dit zeker te weten voeren we een BLAST uit met de geselecteerde exonen en het eerste transcript (zoals getoond in de uitlegvideo). We vinden de volgende resultaten:
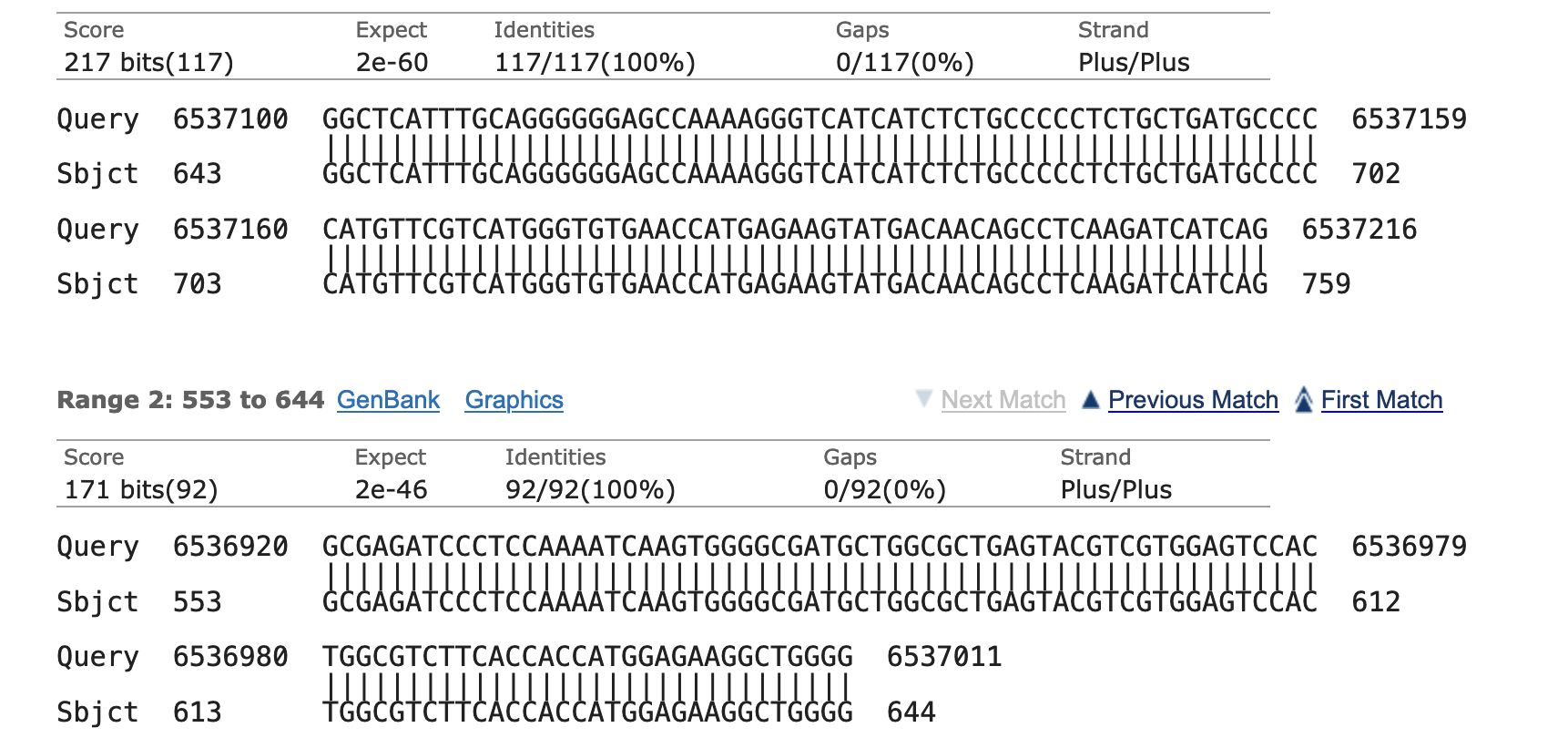 We vinden twee regio’s van overlap. Deze blijken overeen te komen met exon 4 en 5:
We vinden twee regio’s van overlap. Deze blijken overeen te komen met exon 4 en 5:
- Exon 4: 553 t/m 643.
- Exon 5: 644 t/m 759.
qPCR primers ontwerpen met Primer-BLAST
Net als voor ontwerpen van PCR primers, kunnen we voor het ontwerpen van qPCR primers de NCBI tool Primer-BLAST gebruiken. Voor qPCR primers moeten we de volgende gegevens invoeren in Primer-BLAST:
- de identifier van het transcript dat we willen gebruiken als template (de identifier begint met
NM_); - de zoekgebieden voor de forward en de reverse primers (in de praktijk zijn dit coördinaten van de twee opeenvolgende exonen die we hebben uitgekozen);
- en de minimale en maximale PCR productgrootte (80-125 nucleotiden).
In de onderstaande video wordt dit getoond met de eerder gekozen exonen van het MAP2 gen:
De output van Primer-BLAST is een lijst met primerparen die gebruikt kunnen worden voor qPCR. De output bevat de volgende gegevens:
- De lengte van de primers. De lengte moet tussen de 18 en 24 basen zijn.
- De start- en eindpositie van de primers in opgegeven NCBI sequentie.
- De meltingtemperatuur van de primers. Deze temperatuur moet voor de forward en de reverse primers ongeveer hetzelfde zijn.
- Het percentage G en C nucleotiden in de primers. Dit percentage moet tussen de 45-55% liggen.
- Twee getallen die iets zeggen over de vorming van secundaire structuren (self complementarity en self 3’ complementarity). Deze getallen moeten zo laag mogelijk zijn.
qPCR primers controleren met UCSC In Silico PCR
Om meer zekerheid te krijgen over de specificiteit van de qPCR primers, kunnen we de UCSC In Silico PCR tool gebruiken. In de onderstaande video wordt getoond hoe je dat doet:
Voor de UCSC In Silico PCR is het van belang dat je het juiste Target selecteert. Kies je voor het Target ‘genome assembly’ dan controleert de UCSC In Silico PCR tool of de qPCR primers een product kunnen vormen op genomisch DNA. Kies je voor Target ‘GENCODE Genes’ dan controleert de tool of de qPCR primers een product kunnen vormen met transcripten/cDNA. Beide opties kunnen interessant zijn voor het controleren van je qPCR primers.
Net als voor PCR primers geldt voor qPCR primers dat je niet weet of je primers echt goed werken voordat je de primers hebt getest op het lab.
Opdracht 4
Ontwerp qPCR primers voor het VGLUT1 gen m.b.v. Primer-BLAST. Controleer een primerpaar met de UCSC in silico PCR op specificiteit.
Klik hier voor het antwoord
Primer-BLAST vindt meerdere primers. We kiezen een primerpaar die voldoet aan de ontwerpeisen (het achtste primerpaar):

Opdracht 4
Ontwerp qPCR primers voor het GAPDH gen m.b.v. Primer-BLAST. Controleer een primerpaar met de UCSC in silico PCR op specificiteit.
Klik hier voor het antwoord
Tijdens het ontwerpen van de primers geeft Primer-BLAST aan dat er transcripten zijn die overeenkomen met ons transcript:
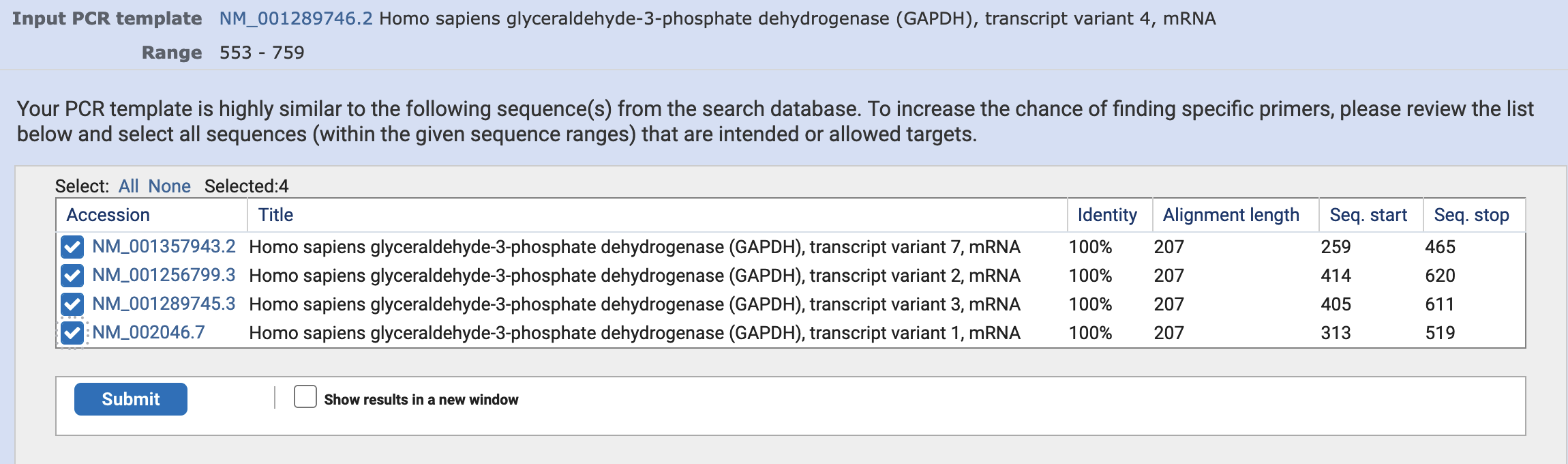 Dit zijn de transcripten die we verwachten, omdat de gekozen exonen ook in deze transcripten aanwezig zijn. Je kunt ervoor kiezen om de transcripten aan te vinken of uitgevinkt te laten.
Dit zijn de transcripten die we verwachten, omdat de gekozen exonen ook in deze transcripten aanwezig zijn. Je kunt ervoor kiezen om de transcripten aan te vinken of uitgevinkt te laten.
Primer-BLAST vindt meerdere primers. We kiezen een primerpaar die voldoet aan de ontwerpeisen (het vierde primerpaar):

Dit primerpaar is volgens de in silico PCR niet specifiek: als we de Target voor de in silico PCR instellen op ‘genome assembly’ vinden we wel (!) een resultaat:
 Voor een aantal andere primerparen in de resultaten (bijvoorbeeld het onderstaande primerpaar) is dit niet het geval. Mogelijk zijn die primerparen dus een betere keuze.
Voor een aantal andere primerparen in de resultaten (bijvoorbeeld het onderstaande primerpaar) is dit niet het geval. Mogelijk zijn die primerparen dus een betere keuze.

Samenvatting: Genexpressie meten
In de onderstaande video wordt een overzicht gegeven van de verschillende stappen die nodig zijn om de genexpressie van een gen met qPCR te meten:
PCR en qPCR verschillen van elkaar op de volgende punten:
- Bij PCR gebruiken we DNA als uitgangsmateriaal (ook voor het ontwerpen van primers, dus een sequentie met een NCBI nucleotide ID die begint met
NG_). Bij qPCR gebruiken we RNA als uitgangsmateriaal (ook voor het ontwerpen van primers, dus een sequentie met een NCBI nucleotide ID die begint metNM_). - Bij PCR is vaak het doel om het gen te kopiëren als startpunt voor een andere analyse, bijvoorbeeld sequencing. Bij qPCR is het doel de genexpressie kwantificeren in een experimentele conditie.
- Bij PCR kijken we alleen naar het gen van interesse (en alleen voor dat gen ontwerpen we primers). Bij qPCR meten we de expressie van het gen van interesse en van een referentiegen, zodat we de resultaten kunnen normaliseren. Voor beide genen moeten primers ontworpen worden.
Werkcollege
Casus Les 4
Mensen met lactose-intolerantie hebben last van spijsverteringsklachten (zoals misselijkheid en diarree) na het eten van melkproducten. Bij deze mensen wordt er onvoldoende werkzaam lactase geproduceerd. Lactase is het enzym dat de suiker lactose in melkproducten kan verteren tot glucose en galactose.
Voer de volgende opdrachten uit voor deze casus:
Het enzym lactase wordt gecodeerd door het gen LCT. Zoek voor dit gen de volgende algemene informatie op:
- Op welk chromosoom is LCT aanwezig?
- Hoeveel exonen heeft LCT?
- In welke weefsels komt LCT vooral tot expressie?
- Wat is de functie van het eiwit waar LCT voor codeert?
In heel zeldzame gevallen komt lactose-intolerantie voor bij baby’s. Hierdoor kunnen ze geen borstmelk verdragen. De oorzaak is dan soms een mutatie in exon 9 van het LCT gen. We willen voor een baby met lactose-intolerantie bepalen of er sprake is van een mutatie in exon 9 van het LCT gen.
Ontwerp PCR primers om Sanger sequencing op exon 9 uit te kunnen voeren. Maak hiervoor gebruik van Primer-BLAST en kies uit de gevonden primers één primerpaar die het beste voldoet aan de ontwerpeisen. Controleer voor deze primer de specificiteit met de in silico PCR tool van UCSC.
De PCR primers worden besteld en blijken in het lab te werken. De primers worden gebruikt om een PCR uit te voeren op het DNA van de patiënt. Vervolgens wordt er Sanger sequencing uitgevoerd op het PCR product. De resultaten zijn de vinden in het FASTA bestand
casus_les04_sangersequencing_patient.fa. Bepaal of er een mutatie aanwezig is in exon 9 van de patiënt en zo ja, wat het effect van de mutatie is op het eiwit?
Voor de meeste mensen met lactose-intolerantie is er geen sprake van een mutatie in het lactase gen. In plaats daarvan komt het gen minder tot expressie. Voor een patiënt willen we de expressie van het LCT gen bepalen in de cellen van een biopt die uit de darm van de patiënt is genomen.
- Ontwerp qPCR primers om de expressie van het LCT gen te kunnen meten. Maak hiervoor gebruik van Primer-BLAST en kies uit de gevonden primers één primerpaar die het beste voldoet aan de ontwerpeisen. Controleer voor deze primer de specificiteit met de in silico PCR tool van UCSC.