Les 5
Leeruitkomsten
- De student kan informatie over posttranslationele modificaties en eiwitdomeinen in een eiwit opzoeken in de NCBI eiwitdatabase.
- De student kan NCBI (protein) BLAST gebruiken om een eiwit te vergelijken met andere bekende eiwitten in een bepaald organisme.
Voorbereiding
Biologische achtergrondinformatie
De primaire eiwitstructuur
Om de genetische boodschap van een gen te vertalen naar een eiwit moet het gen eerst worden gekopieerd naar mRNA (transcriptie) en vervolgens moet het mRNA door de ribosomen worden vertaald naar een keten van aminozuren (translatie). Hiervoor binden de ribosomen aan het startcodon op het mRNA en zetten vervolgens elk codon van de coderende sequentie (CDS) om naar een aminozuur. Dit proces stopt zodra de ribosomen een stopcodon tegenkomen. Op dat moment laten de ribosomen het mRNA en de geproduceerde aminozuurketen los. De gevormde aminozuurketen is de primaire eiwitstructuur.
De primaire eiwitstructuur van alle eiwitten in de natuur bestaat slechts uit twintig aminozuren. Die aminozuren zijn te verdelen in vier verschillende groepen:
- Hydrofobe aminozuren: alanine (A), glycine (G), isoleucine (I), leucine (L), methionine (M), fenylalanine (F), proline (P), tryptofaan (W) en valine (V).
- Polaire (ongeladen) aminozuren: asparagine (N), cysteine (C), glutamine (Q), serine (S), threonine (T) en tyrosine (Y).
- Zure (negatief geladen) aminozuren: asparaginezuur (D) en glutaminezuur (E).
- Basische (positief geladen) aminozuren: arginine (R), histidine (H) en lysine (K).
De primaire eiwitstructuur bepaalt de eiwitvouwing en dus de functie van een eiwit.
Posttranslationele modificaties
Nadat de primaire eiwitstructuur is gevormd door de ribosomen, kan het eiwit gemodificeerd worden. Dit gebeurt veelal in het endoplasmatisch reticulum en in het Golgi apparaat, maar kan ook later plaatsvinden (bijvoorbeeld als onderdeel van een signaaltransductieketen). Er zijn verschillende posttranslationele modificaties mogelijk. Een paar voorbeelden:
- Glycosylatie: hierbij wordt een suikergroep gekoppeld aan het eiwit. Suikergroepen bestaan uit verschillende hydrofiele groepen; glycosylering van een eiwit vergroot daarom de oplosbaarheid van het eiwit.
- Fosforylatie: hierbij wordt een fosfaatgroep gekoppeld aan een serine, threonine of een tyrosine. Deze aminozuren zijn ongeladen en de fosforylering geeft ze een negatieve lading.
- Acetylatie: hierbij wordt een acetylgroep (COCH3) gekoppeld aan een lysine. Lysine is meestal positief geladen. De acytylatie zorgt ervoor dat deze positieve lading verdwijnt en dat de lysine ongeladen wordt.
Posttranslationele modificaties geven eiwitten andere chemische eigenschappen (lading en oplosbaarheid). Ook beïnvloeden de modificaties de vorm van het eiwit en de binding aan andere moleculen (bijvoorbeeld andere eiwitten of het DNA).
Eiwitdomeinen
Uiteindelijk vouwt de aminozuurketen zich tot een driedimensionaal, functioneel eiwit. Dit is de tertiaire structuur van het eiwit. In de tertaire structuur zijn één of meerdere functionele onderdelen te herkennen. Dit zijn de eiwitdomeinen. De eiwitdomeinen zijn vaak evolutionair geconserveerd en hebben een specifieke functie. Zo hebben transcriptiefactoren vaak een eiwitdomein wat aan het DNA kan binden en receptoren hebben een eiwitdomein wat aan het ligand kan binden. Enzymen hebben catalytische eiwitdomeinen die een specifieke chemische reactie katalyseren.
De NCBI eiwitdatabase
De NCBI eiwitdatabase is de tegenhanger van de NCBI nucleotidedatabase. Voor elk transcript dat aanwezig is in de NCBI nucleotide database, is er een bijbehorend eiwit in de NCBI eiwitdatabase. De NCBI eiwitdatabase bevat de aminozuurvolgorde (de primaire eiwitstructuur) die hoort bij de coderende sequentie van het transcript.
In de onderstaande video wordt de eiwitdatabase gedemonstreerd voor het HBB eiwit:
Opdracht 5
SMAD2 is een eiwit dat de celdeling remt. Mutaties in het SMAD2 gen kunnen leiden tot kanker.
Zoek in de NCBI database het eiwit op dat hoort bij transcript NM_001003652.4. Wat is de identifier van dit eiwit? En hoe lang is dit eiwit (aantal aminozuren)?
Klik hier voor het antwoord
De identifier is NP_001003652.1.

De lengte van dit eiwit is 467 aminozuren.

Posttranslationele modificaties in de eiwitdatabase
In de eiwitdatabase kun je voor een bepaald eiwit de bekende posttranslationele modificaties vinden. In de onderstaande video wordt dit getoond voor het HBB eiwit:
Opdracht 5
Bepaal voor het SMAD2 eiwit dat je hebt gevonden in de vorige opdracht:
- Hoeveel aminozuren in het eiwit gefosforyleerd kunnen worden?
- Welke aminozuren er gefosforyleerd worden?
Klik hier voor het antwoord
11 aminozuren kunnen worden gefosforyleerd. Dit zijn threonines en serines.Eiwitdomeinen voor een eiwit identificeren
Via de pagina van het HBB eiwit kunnen we ook de domeinen vinden die zijn gevonden voor dit eiwit, zoals in de onderstaande video wordt getoond:
De eiwitdomeinen die je ziet zijn domeinen die voorspeld zijn op basis van een alignment met vergelijkbare domeinen in andere eiwitten. Of de voorspelling waarschijnlijk is, wordt weergegeven met de E-score. Hoe kleiner de E-score is, hoe waarschijnlijker het is dat het gevonden domein ook daadwerkelijk een domein is in het eiwit.
Opdracht 5
Bepaal voor het eerder gevonden SMAD2 eiwit:
- Welke domeinen aanwezig zijn in dit eiwit?
- Wat de locaties zijn van deze domeinen in het eiwit?
Klik hier voor het antwoord
In het eiwit zijn waarschijnlijk twee domeinen aanwezig (de E-scores zijn erg laag!):
- Het
MH1_SMAD_2_3domein, dat loopt van aminozuur 8 t/m 172. - Het
MH2_SMAD_2_3domein, dat loopt van aminozuur 266 t/m 456.

Vergelijkbare eiwitten vinden met protein BLAST
Soms wil je voor een eiwit weten of er in hetzelfde organisme vergelijkbare (homologe) eiwitten zijn. We kunnen deze eiwitten identificeren door een protein BLAST uit te voeren. In de onderstaande video wordt getoond hoe je dat doet:
Om de BLAST uit te voeren is het belangrijk dat je op de volgende zaken let:
- Zorg ervoor dat je de blastp BLAST geselecteerd hebt. Dit is namelijk het BLAST algoritme wat je gebruikt om aminozuursequenties met elkaar te vergelijken.
- Voer de identifier van jouw eiwit (de identifier begint met
NP_) in in het vak voor de query sequentie. - Selecteer de eiwitdatabase die je wilt gebruiken, bijvoorbeeld
refseq_select. - Selecteer de diersoort waarin je de homologen wilt zoeken. Vaak is dat de mens (
Homo sapiens).
Voor de gevonden resultaten worden een aantal parameters gegeven, waarvan de volgende belangrijk zijn:
- Query coverage: hoeveel procent van de query lengte heeft overlap met het gevonden eiwit? Stel dat ons eiwit 100 aminozuren is en het gevonden eiwit slechts 50 aminozuren, maar dat die 50 aminozuren wel helemaal overlappen met ons eiwit. In dat geval is de query coverage 50%, omdat 50% van ons eiwit wordt ‘bedekt’ door het gevonden eiwit.
- Percent identity: hoeveel procent van de aminozuren in de overlappende regio zijn hetzelfde? Voor ons eiwit en het gevonden eiwit is een alignment uitgevoerd. Voor de regio die aanwezig is in de alignment (de overlap tussen de twee eiwitten) wordt bepaalt hoeveel procent van de aminozuren hetzelfde zijn in de twee eiwitten (dus hoeveel procent matches er zijn in de alignment).
- E-score: dit geeft de significantie van de vondst aan. Hoe kleiner de E-score, hoe waarschijnlijker het gevonden eiwit significante overlap heeft (en dus homologie vertoont) met ons eiwit.
Opdracht 5
Bepaal voor het eerder gevonden SMAD2 eiwit:
- Welke eiwit in de mens het meeste overeenkomt met het SMAD2 eiwit?
- Welk deel van het SMAD7 eiwit (welke aminozuurposities) overlappen met het SMAD2 eiwit?
Klik hier voor het antwoord
Het eiwit dat het meeste lijkt op SMAD2 is SMAD3:
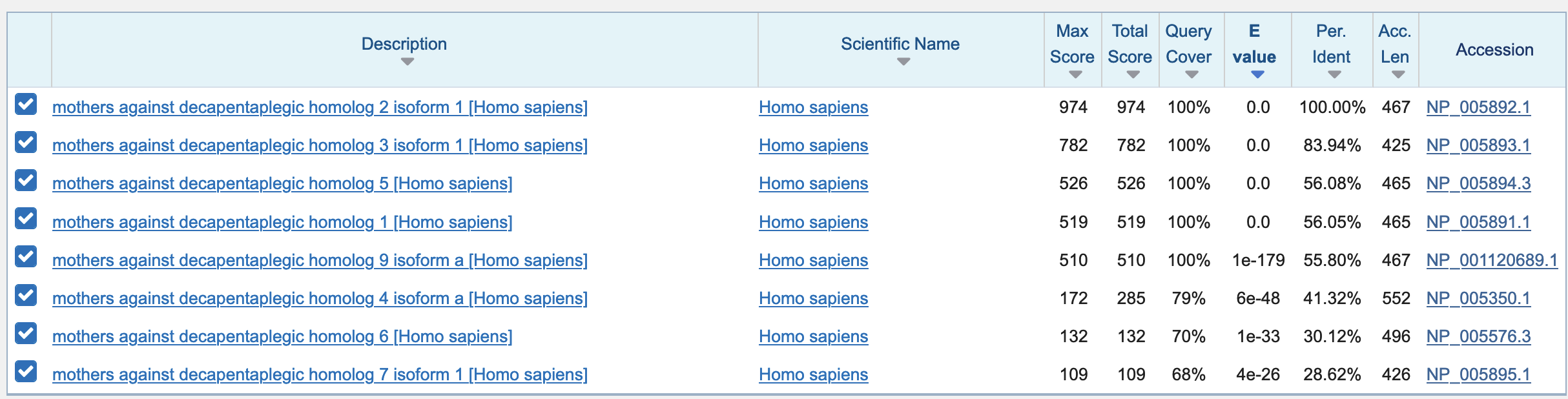
NB: de gevonden eiwitten worden automatisch gesorteerd op de E-score. Voor SMAD3 is de E-score het laagste en het percentage identieke aminozuren het hoogst. Het eerste eiwit is natuurlijk SMAD2 zelf!
SMAD7 overlapt van aminozuur 157 t/m 424 met SMAD2:
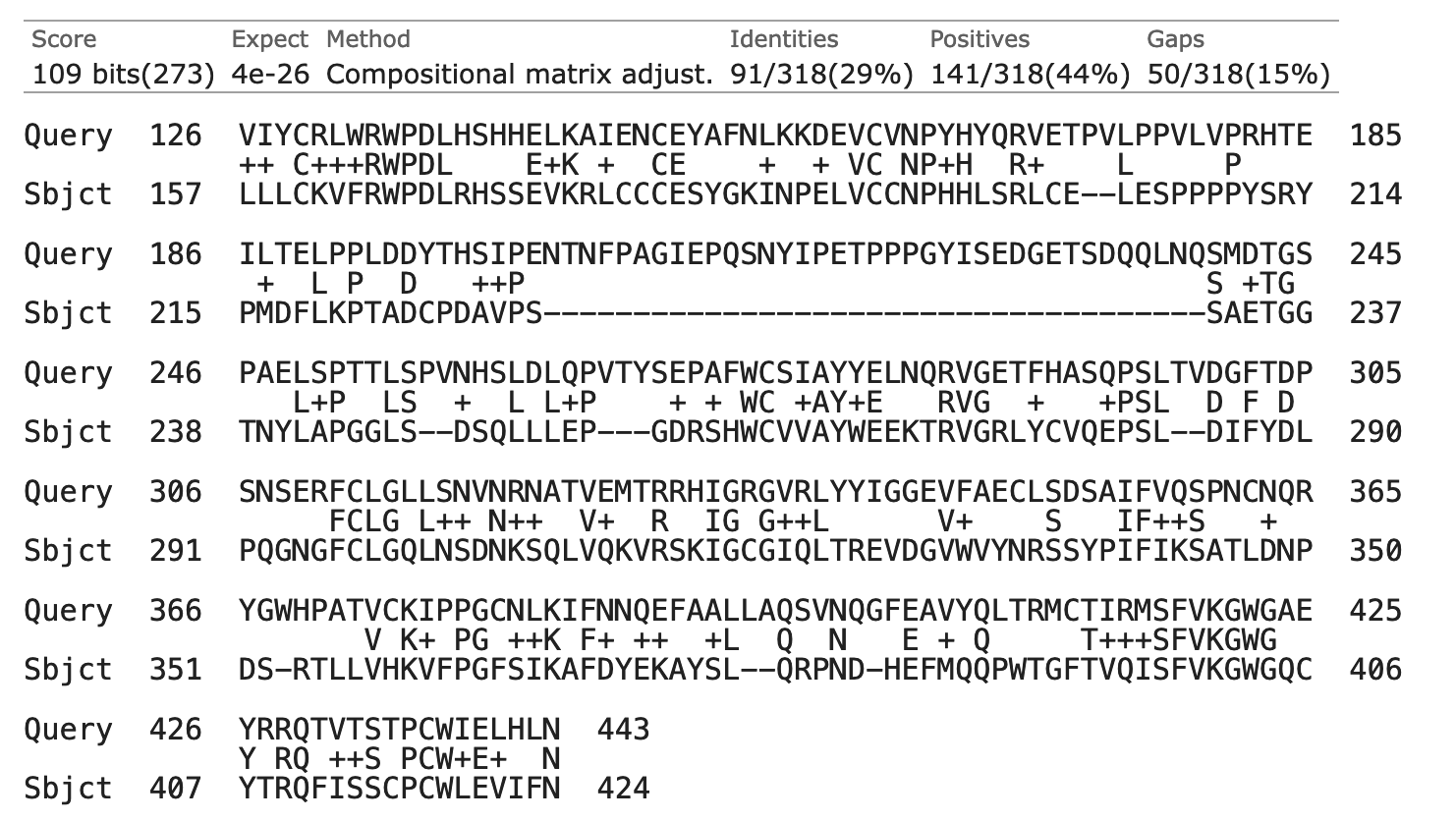
Werkcollege
Casus Les 5
In verschillende kankersoorten is er sprake van een defect in het TP53 eiwit. Normaal gesproken remt dit eiwit de deling van cellen. Als het eiwit niet meer goed werkt, kan dat leiden tot ongecontroleerde celdeling en zo uiteindelijk tot de vorming van een tumor.
Voor een kankerpatiënt is een biopt genomen van de tumor. De artsen verwachten dat het eiwit TP53 een rol speelt in deze tumor. Ze willen dit onderzoeken.
Voer de volgende opdrachten uit voor deze casus:
Zoek algemene informatie op voor het TP53 gen:
- Op welk chromosoom is TP53 aanwezig?
- Hoeveel exonen heeft TP53?
- In welke weefsels komt TP53 vooral tot expressie?
- Wat is de functie van het eiwit TP53?
- De artsen willen onderzoeken of het TP53 gen normaal tot expressie komt. Ze willen hiervoor de expressie meten met qPCR. Ontwerp qPCR primers om de expressie van het TP53 gen te kunnen meten. Maak hiervoor gebruik van Primer-BLAST en kies uit de gevonden primers één primerpaar die het beste voldoet aan de ontwerpeisen. Controleer voor deze primer de specificiteit met de in silico PCR tool van UCSC.
De expressie van het TP53 gen blijkt normaal te zijn. De onderzoekers willen weten of er sprake is van een mutatie in het TP53 gen en voeren hiervoor een Sanger sequencing uit. Er wordt een mutatie gevonden in exon 4 van het TP53 gen.
De resultaten van de Sanger sequencing zijn de vinden in het FASTA bestand
casus_les05_sangersequencing_patient.fa. Bepaal voor deze mutatie:- wat het effect is op het eiwit (gebruik voor het detecteren van de mutatie transcript 1 van het TP53 gen);
- of het gemuteerde residu een plaats is voor posttranslationele modificaties en zo ja, wat het effect is van de mutatie voor die modificatie;
- en in welk eiwitdomein de mutatie valt en wat dit betekent voor het mogelijke effect van de mutatie.