Les 6
Leeruitkomsten
- De student kan informatie over een eiwit opzoeken in de Uniprot database.
- De student kan het moleculaire gewicht van een eiwit opzoeken (bijvoorbeeld voor Western blot analyses) in de Uniprot database.
- De student kan voor een eiwit de eiwitstructuur opzoeken in de Uniprot database en hierbij onderscheid maken tussen experimenteel bepaalde en computationeel voorspelde eiwitstructuren.
Voorbereiding
Biologische achtergrondinformatie
De tertaire structuur van een eiwit bepalen
De aminozuurketen die op basis van de instructies in het mRNA door de ribosomen is gevormd, zal zich in de cel vouwen tot een functioneel eiwit. De vorm van het eiwit die door de vouwing ontstaat is de tertiaire structuur. Om te begrijpen hoe een eiwit werkt, is het noodzakelijk om de tertiaire structuur van het eiwit te kennen. Er zijn verschillende experimentele methoden om de tertiaire structuur te bepalen, waaronder Röntgen kristallografie, NMR en elektronenmicroscopie. De meeste experimenteel bepaalde structuren zijn bepaald met Röntgen kristallografie. Bij deze techniek wordt het eiwit in kristalvorm gebracht (dit is een erg tijdrovende klus). Het kristal wordt vervolgens bestraald met Röntgenstralen. Als de Röntgenstralen door het kristal gaan, zullen ze door de atomen van de eiwitten in het kristal worden afgebogen. Op basis van het buigingspatroon kan worden berekend wat de driedimensionale vorm van het eiwit is.
Het experimenteel bepalen van de tertiaire structuur is een tijdrovende klus, maar tot voor kort was dit wel de enige manier om de vorm van het eiwit te bepalen. Er waren wel computermodellen die op basis van de aminozuursequentie van het eiwit (de primaire structuur) de vorm van het eiwit konden voorspellen, maar de voorspellingen waren niet zo goed. Echter, een paar jaar geleden zijn er modellen ontwikkeld die gebruik maken van artificiële intelligentie. Het bekendste model en meest succesvolle model is Alphafold. Alphafold blijkt met een hoge precisie de structuur van eiwitten te kunnen voorspellen op basis van de aminozuursequentie. Alphafold is getraind met alle bekende (experimenteel bepaalde) eiwitstructuren: door deze structuren en hun aminozuursequentie aan te bieden aan Alphafold en de voorspellingen van het algoritme te ‘corrigeren’, heeft het algoritme geleerd om nauwkeurige voorspellingen te maken. Een voorbeeld van een voorspelling is te zien in het onderstaande plaatje.
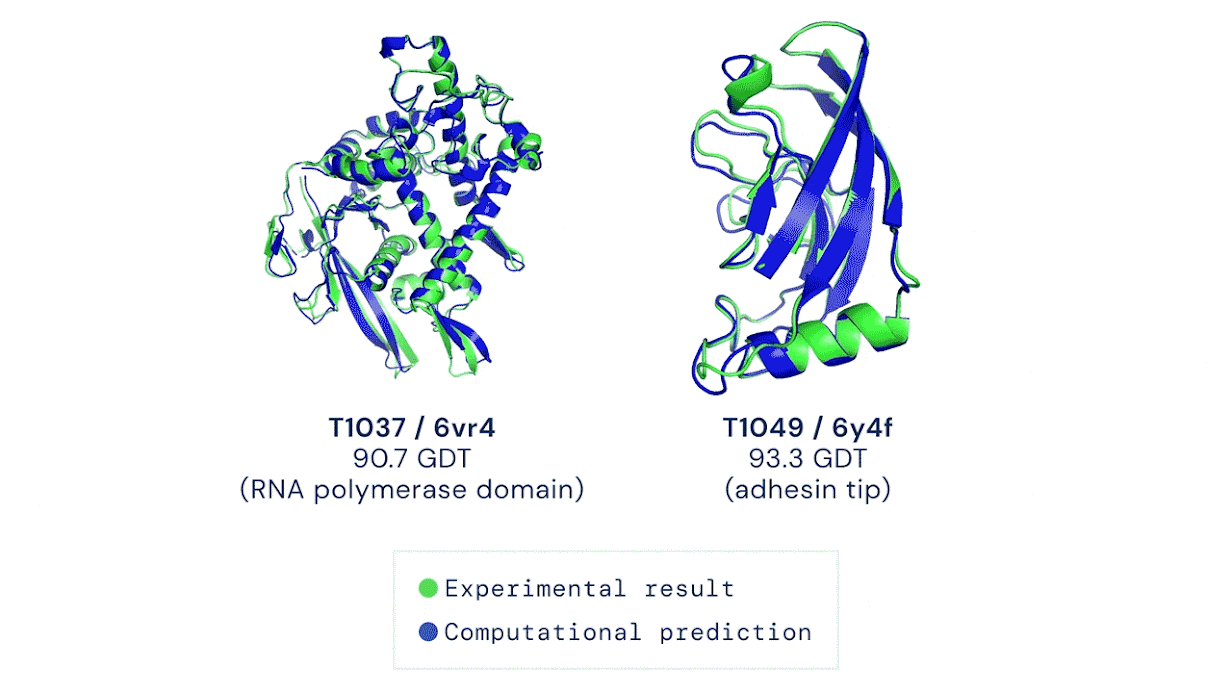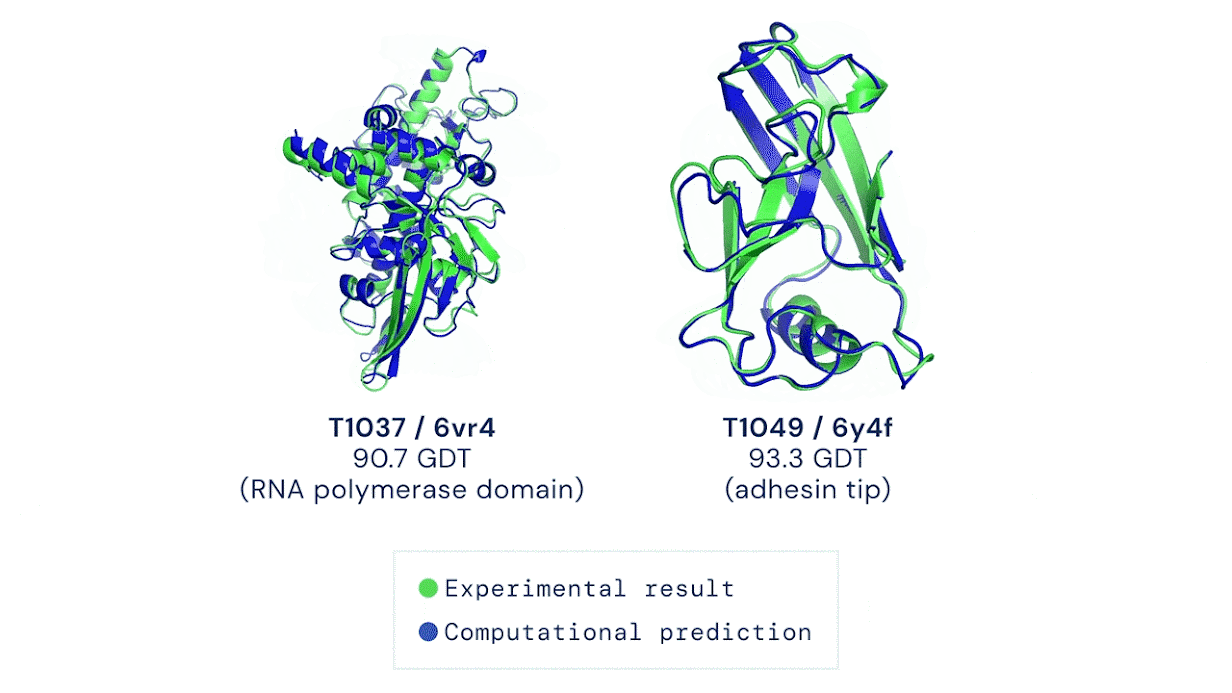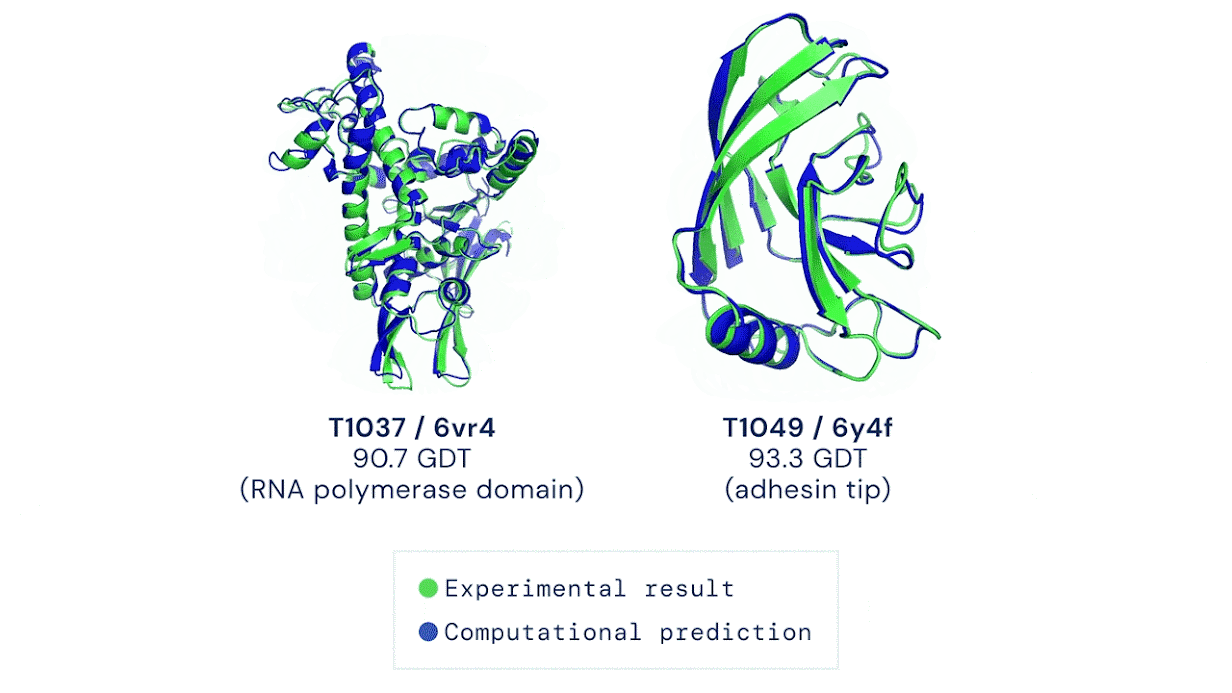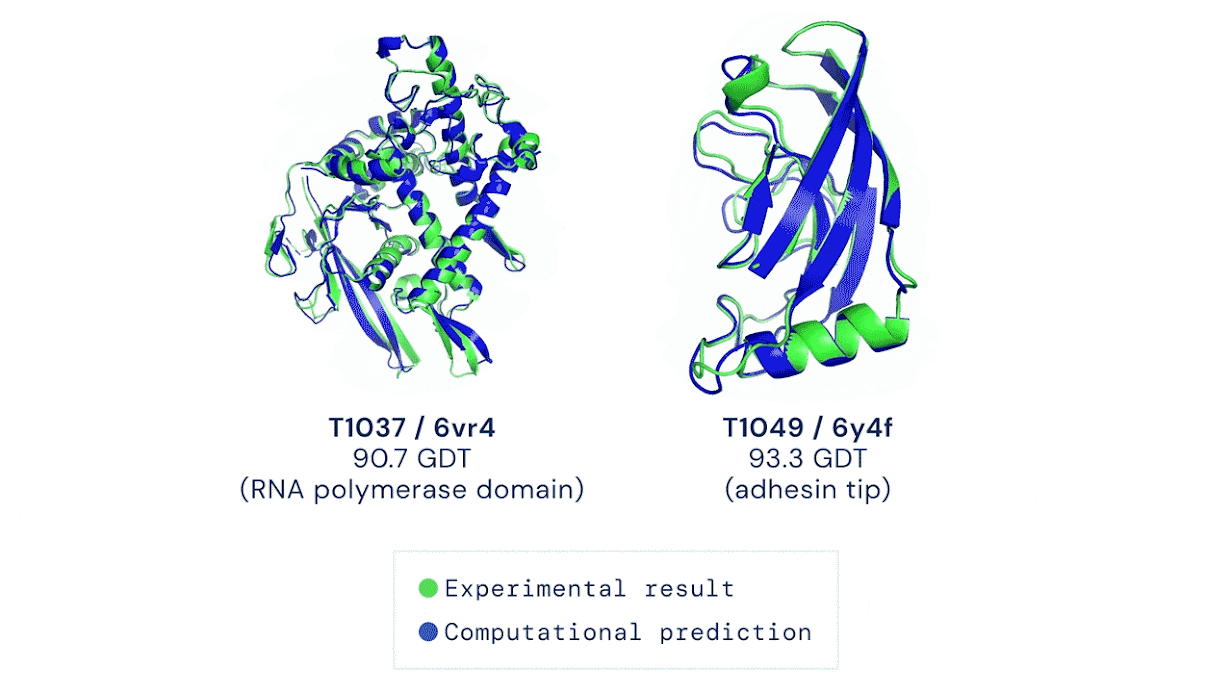
Hoewel de komst van Alphafold en de opvolgers van dit algoritme enorm veel nieuwe mogelijkheden hebben gebracht voor het onderzoeken van eiwitten (en bijvoorbeeld ook voor het ontwikkelen van medicijnen), kunnen we niet genoeg benadrukken dat het hier gaat om voorspellingen. Dit betekent dat we niet zeker weten of de eiwitstructuren die Alphafold gevonden heeft ook daadwerkelijk kloppen. Het blijft noodzakelijk om de structuur experimenteel te bepalen om hierover volledige zekerheid te krijgen.
Eiwit-eiwitinteracties
Eiwitten werken vaak samen om een bepaalde functie uit te voeren. Hiervoor gaan eiwitten interacties met elkaar aan. Het kan zijn dat de eiwitten hiervoor een complex vormen dat bestaat uit meerdere exemplaren van hetzelfde eiwit of uit exemplaren van verschillende eiwitten. Deze complexen vormen de quaternaire eiwitstructuren. Maar niet alle eiwitinteracties zijn langdurig zoals in eiwitcomplexen. Soms is er sprake van een korte interactie, zoals in een signaaltransductieketen: in dat geval leidt de eiwitinteractie tot de activering of remming van het volgende eiwit in de keten.
Informatie over eiwitten opzoeken in Uniprot
In de vorige lessen hebben we informatie over genen, transcripten en eiwitten opgezocht in de NCBI database. Er zijn nog veel meer andere databases waarin informatie over genen en eiwitten is opgeslagen. Eén van de bekendste en meest gebruikvriendelijke databases voor eiwitten is de Uniprot database. In de Uniprot database is het uitgangspunt voor je zoektocht het eiwit waarin je geïnteresseerd bent. Voor een eiwit kun je verschillende informatie opzoeken, zoals de functie, de expressie in het lichaam, de interactie met andere eiwitten en ziektes waarbij het eiwit betrokken is.
In de onderstaande video wordt getoond hoe je algemene informatie over een eiwit kunt vinden in de Uniprot database. Net als in de vorige les kijken we hiervoor naar het voorbeeld van het HBB eiwit:
Ziektes en ziekteveroorzakende mutaties opzoeken in Uniprot
Mutaties in een gen leiden soms tot aminozuurveranderingen in het eiwit en deze aminozuurveranderingen kunnen de structuur en functie van het eiwit verstoren. Op die manier kunnen mutaties leiden tot ziektes. Veel van de ziekteveroorzakende mutaties zijn te vinden in de Uniprot database. In de onderstaande video wordt getoond hoe je voor een eiwit de ziektes en ziekteveroorzakende mutaties kunt opzoeken in Uniprot:
Eiwitinteracties opzoeken in Uniprot
Of een eiwit functioneert in een complex of andere interacties aangaat met eiwitten, kun je opzoeken in de ‘Interactions’ sectie van de Uniprot database. In de onderstaande video wordt getoond hoe je dat doet:
Opdracht 6
In de vorige les hebben we informatie opgezocht voor het SMAD2 eiwit in de NCBI database. Nu gaan we informatie opzoeken in de Uniprot database. Zoek voor het SMAD2 eiwit de volgende zaken op:
In welke weefsels/cellen komt SMAD2 tot expressie?
Mutaties in SMAD2 kunnen aangeboren hartafwijkingen veroorzaken (‘congenital heart disease’). Welke mutaties zijn er voor SMAD2 geassocieerd met deze ziekte?
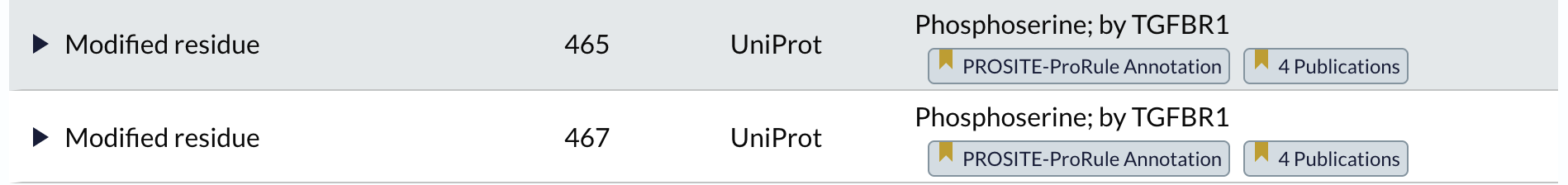
SMAD2 wordt (onder andere) gefosforyleerd door TGFBR1. Welke residuen in SMAD2 worden door TGFBR1 gefosforyleerd?
Met welke andere eiwitten vormt SMAD2 een complex?
Klik hier voor het antwoord
- SMAD2 komt tot expressie in spierweefsel, het hart, endotheelcellen en de placenta.

- De volgende mutaties in SMAD2 kunnen leiden tot aangeboren hartafwijkingen:

- TGFBR1 fosforyleert de volgende residuen in SMAD2:
- SMAD2 vormt een complex met SMAD3, SMAD4 en mogelijk ook met TRIM33 en ZNF451.

Het moleculaire gewicht en de aminozuursequentie opzoeken in Uniprot
Voor sommige experimenten op het lab moet je het moleculaire gewicht van een eiwit weten. Een voorbeeld hiervan is Western blot. Je kunt met behulp van de aminozuursequentie het moleculaire gewicht zelf berekenen. Het alternatief is om het gewicht op te zoeken in de Uniprot database. In de onderstaande video wordt getoond hoe je dat doet:
Opdracht 6
Wat is het moleculaire gewicht van (de eerste isoform van) het SMAD2 eiwit?
Klik hier voor het antwoord
52 kDa:

De eiwitstructuur opzoeken in Uniprot
In de Uniprot database kun je ook alle experimenteel bepaalde structuren vinden voor een eiwit. Ook de door Alphafold voorspelde structuur is opgenomen op de Uniprot pagina. In het overzicht kun je zien in welke database de eiwitstructuur te vinden is. Voor experimenteel bepaalde structuren is dat meestal de Protein Data Bank. Verder kun je in het overzicht vinden welke methode is gebruikt om de structuur van het eiwit te bepalen: Röntgen kristallografie (‘X-ray’), NMR, elektronenmicroscopie (‘EM’) of Alphafold (‘predicted’). Voor de experimenteel bepaalde structuren geldt verder dat er is aangegeven met welke resolutie de structuur is bepaald. De resolutie is weergegeven in Ångström (1 Å = 0,1 nm = 1 x 10-10 m).
In de onderstaande video wordt uitgelegd hoe je de eiwitstructuren kunt raadplegen:
Opdracht 6
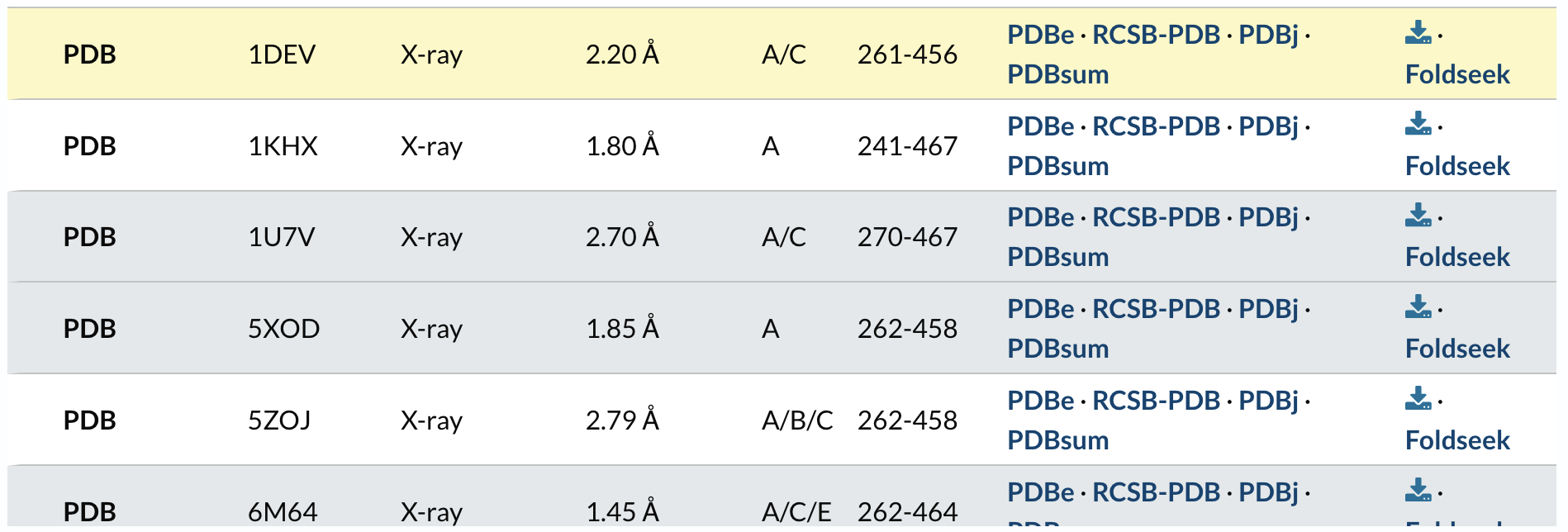
Voor SMAD2 zijn er meerdere experimenteel bepaalde structuren beschikbaar, maar voor al die structuren geldt dat de structuur voor slechts een deel van het eiwit is bepaald. Welk deel van het SMAD2 eiwit is dat meestal?
Klik hier voor het antwoord
Voor de meeste structuren geldt dat aminozuur 262 t/m 467 in kaart zijn gebracht:
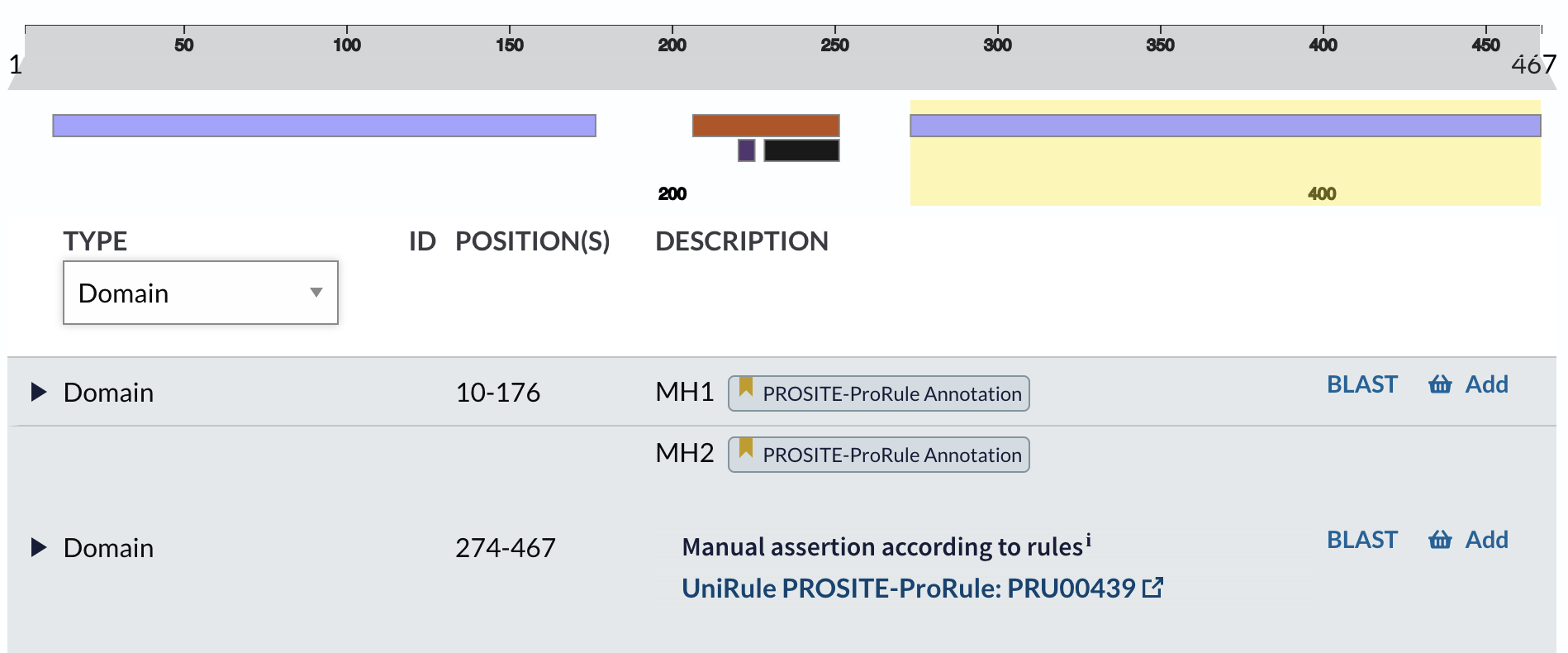
Dit is het MH2 domein van SMAD2:
Werkcollege
Casus Les 6
In de casus van les 3 heb je een mutatie in het ALK eiwit gevonden. In deze les gaan we deze mutatie verder onderzoeken met de Uniprot database.
Voer de volgende opdrachten uit voor deze casus:
Het ALK eiwit is een receptor tyrosine kinase. Wat weet je al over dit type receptor uit eerdere cursussen?
Zoek algemene informatie op over ALK in de Uniprot database:
- In welke weefsels komt ALK vooral tot expressie?
- Wat is de functie van het ALK eiwit?
- Komt de gevonden informatie overeen met de informatie die je in les 3 hebt verkregen uit de NCBI database?
Vormt ALK een complex? (Met andere woorden: is er een quaternaire structuur voor ALK bekend?) Sluit dit aan bij je verwachtingen?
Is de mutatie die je in les 3 hebt gevonden ook terug te vinden in de ‘Disease & Variants’ sectie van ALK pagina in de Uniprot database?
In welk domein van het ALK eiwit is de mutatie aanwezig? Wat is de functie van dat domein?
Voor ALK zijn meerdere experimenteel bepaalde structuren beschikbaar voor het domein waar de mutatie in zit. Er is zelfs een structuur beschikbaar voor het eiwit met de mutatie! Deze structuur heeft PDB identifier ‘4FNX’. De structuur van het wildtype eiwit heeft PDB identifier ‘3L9P’. Zoek in beide structuren het aminozuur op dat gemuteerd is en bekijk de verschillen in structuur op die plek. Wat valt je op?
We willen een Western blot uitvoeren met het ALK eiwit. We gebruiken hiervoor de volgende eiwitmarker:
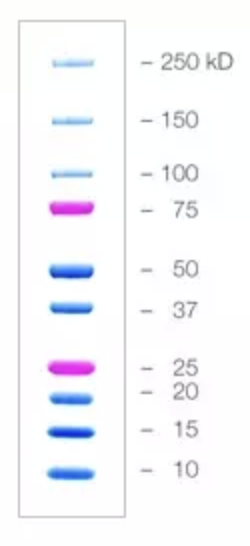
Ter hoogte van welke bandjes van de marker verwacht je het bandje van het ALK eiwit op de Western blot?