Les 7
Leeruitkomst
De student kan netwerkkaarten opzoeken in de KEGG database en de netwerkkaarten interpreteren.
Voorbereiding
Biologische achtergrondinformatie
In de afgelopen lessen hebben we gekeken naar individuele genen en eiwitten. Maar in een biologisch systeem, zoals een cel, werken alle eiwitten samen om leven mogelijk te maken. Uiteindelijk willen we dan ook begrijpen hoe al die verschillende eiwitten samenwerken. De systeembiologie is het vakgebied binnen de biologie die zich hiermee bezig houdt. Systeembiologen proberen alle bekende biologische informatie over genen en eiwitten te integreren in modellen. Met deze modellen kunnen we beter begrijpen hoe een verstoring van een eiwit (bijvoorbeeld door een mutatie) leidt tot een verstoring van het systeem en zo tot ziekte.
De modellen die systeembiologen gebruiken kunnen complex zijn en ook veel wiskunde bevatten. Het ultieme doel van de systeembiologie is om de processen die zich in de cel of in een organisme afspelen in de computer na te bootsen. Een eerste stap in het maken van modellen is de relaties tussen verschillende componenten van het systeem (vaak eiwitten, maar ook bijvoorbeeld DNA en andere biomoleculen) vast te leggen in een netwerk. Voorbeelden van deze relaties zijn jullie al eerder tegengekomen. Zo hebben jullie kennis gemaakt met metabole routes zoals de glycolyse en de citroenzuurcyclus. In deze metabole routes werken enzymen samen om glucose af te breken. Verder hebben jullie kennis gemaakt met signaaltransductieroutes, waarin verschillende eiwitten het signaal van bijvoorbeeld een hormoon, doorgeven in de cel om zo een reactie in de cel te veroorzaken.
Door biologische processen vast te leggen in netwerken, kunnen we eenvoudiger zien wat er gebeurt als een bepaald eiwit niet meer goed functioneert. In het netwerk kunnen we dan immers nagaan met welke andere eiwitten het eiwit samenwerkt en zo kunnen we zien welke routes in het netwerk verstoord worden. Deze informatie is niet alleen nuttig voor het begrijpen van verstoringen (zoals bij mutaties), maar ook om medicijnen te ontwikkelen die de verstoringen ongedaan kunnen maken.
Netwerkinformatie opzoeken in de KEGG database
Een van de belangrijkste databases waarin biologische netwerken zijn vastgelegd is de KEGG database. De KEGG database bevat interactieve netwerkkaarten waarin de relaties tussen eiwitten zijn vastgelegd. Er zijn voor heel veel biologische processen netwerkkaarten beschikbaar. In de onderstaande video wordt getoond hoe je informatie in de KEGG database kunt opzoeken:
Netwerkkaarten in de KEGG database interpreteren
In de netwerkkaarten van de KEGG database is heel veel informatie te vinden. Veel elementen in de kaarten zijn ook interactief en weer verbonden met andere objecten in de database. In de onderstaande video wordt getoond hoe je de netwerkkaarten van de KEGG database kunt interpreteren:
Bovenaan elke netwerkkaart in de KEGG database is een ‘help’ link aanwezig. Door op deze link te klikken kun je de legenda van de kaarten zichtbaar maken. De legenda is ook te bereiken via deze link.
Opdracht 7
Zoek de netwerkkaart op voor cAMP signalering (‘cAMP signaling pathway’). PKA fosforyleert verschillende eiwitten, waaronder NF\(\kappa\)B en NF-AT. Is het effect van deze fosforylaties hetzelfde?
Klik hier voor het antwoord
Nee. Bij NF\(\kappa\)B leidt de fosforylatie tot activatie, terwijl bij NF-AT de fosforylatie leidt tot inactivatie.Opdracht 7
Zoek de netwerkkaart op voor TGF\(\beta\) signalering (‘TGF-beta signaling pathway’). Zoek in de netwerkkaart de cellocatie op voor de volgende eiwitten:
- TGF\(\beta\)
- TGF\(\beta\)RI
- SMAD4
- Het SMAD2/3/4 complex
Klik hier voor het antwoord
- Buiten de cel.
- In het celmembraan.
- In het cytoplasma.
- In de celkern (het complex vormt zich in het cytoplasma en migreert daarna naar de celkern).
Opdracht 7
We gaan verder met de netwerkkaart voor TGF\(\beta\) signalering (‘TGF-beta signaling pathway’). In deze kaart is te zien dan SMAD2, SMAD3 en SMAD4 een complex vormen en via andere eiwitten de genexpressie beïnvloeden. Via welke eiwitten stimuleert het SMAD2/3/4 complex de genexpressie? En via welke eiwitten leidt het SMAD2/3/4 complex juist tot remming van de genexpressie?
Klik hier voor het antwoord
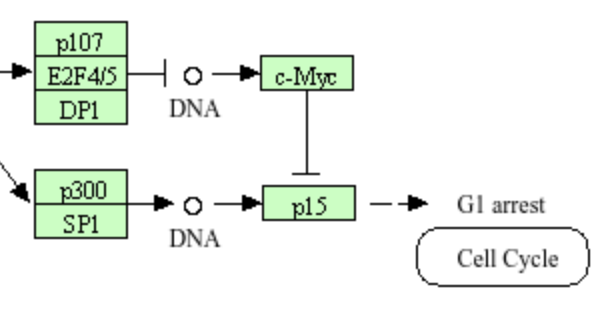
- Activering van p107, E2F4/5 en DP1 leidt tot het remmen van de genexpressie.
- Activering van p300 en SP1 leidt tot het stimuleren van de genexpressie.
Ziektegerelateerde processen opzoeken in de KEGG database
De KEGG database bevat ook een aantal netwerkkaarten voor ziektes. In deze kaarten zijn alle processen weergegeven die een rol spelen bij de ziekte en die verstoord zijn bij de ziekte. In de onderstaande video wordt getoond hoe je de informatie in de ziektegerelateerde netwerkkaarten kunt interpreteren:
Opdracht 7
Gebruik de netwerkkaart voor ‘colorectal cancer’ om de volgende vragen te beantwoorden:
- Wat is de relatie tussen RAF en MEK?
- Waar in de cel is TP53 (p53) actief?
- Is SMAD2 een tumor suppressor gen of een oncogen?
Klik hier voor het antwoord
- Raf activeert MEK door MEK te fosforyleren.
- In de celkern.
- Een tumor suppressor gen: kanker kan ontstaan als SMAD2 niet meer goed functioneert.
Opdracht 7
Zoek de netwerkkaart op voor AML (‘acute myeloid leukemia’). Is de receptor FLT3 een oncogen of een tumor suppressorgen? Betekent dat dat een mutatie in FLT3 leidt tot een overactief eiwit of juist tot een inactief eiwit?
Klik hier voor het antwoord
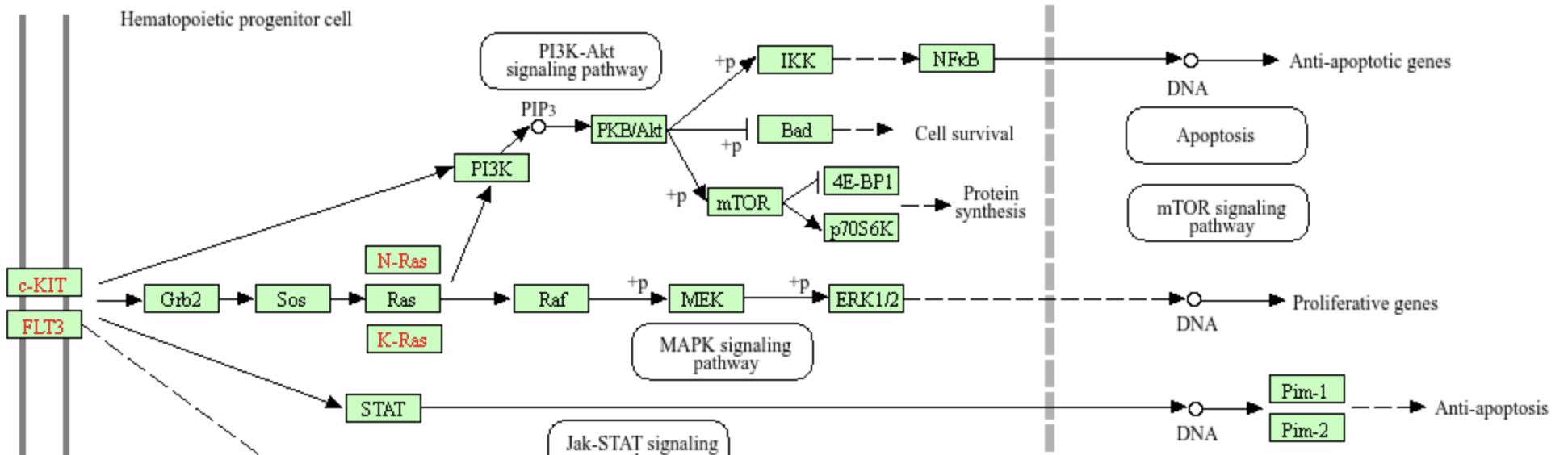
Volgens het overzicht in KEGG is FLT3 een oncogen. Bovendien stimuleert FLT3 processen die te maken hebben met celdeling en het voorkomen van celdood (apoptose). Dit betekent dat het eiwit in gemuteerde vorm actiever moet zijn om kanker te veroorzaken, want bij kanker is juist sprake van meer celdeling en minder apoptose.
Werkcollege
Casus Les 7
Voor een patiënt met maagkanker is een biopt genomen van de tumor. De artsen verwachten dat het eiwit KRAS een rol speelt in deze tumor en ze willen dit onderzoeken.
Voer de volgende opdrachten uit voor deze casus:
Zoek algemene informatie op over KRAS in de Uniprot database:
- Wat is de functie van het KRAS eiwit?
- Wat is de lengte van het KRAS eiwit?
- Wat is het moleculaire gewicht van het KRAS eiwit?
Er wordt Sanger sequencing uitgevoerd op exon 2 van het KRAS gen. De resultaten zijn de vinden in het FASTA bestand
casus_les07_sangersequencing_patient.fa. Bepaal of er een mutatie aanwezig is in exon 2 van de patiënt en zo ja, wat het effect van de mutatie is op het eiwit?Is de mutatie ook terug te vinden in de ‘Disease & Variants’ sectie van KRAS pagina in de Uniprot database?
Het KRAS eiwit speelt een rol in een signaaltransductieketen. In de geactiveerde vorm is het eiwit gebonden aan GTP. Het eiwit kan zichzelf inactiveren door het GTP om te zetten naar GDP. Het gemuteerde eiwit van de patiënt blijkt een veel lagere GTPase activiteit te hebben en blijft dus veel langer actief dan het normale eiwit.
Zoek in de KEGG database de pathwaykaart op die hoort bij maagkanker (‘Gastric cancer’). Zoek in deze route KRAS op en beantwoordt de volgende vragen:
- Door welk eiwit wordt KRAS geactiveerd?
- Welk eiwit wordt door KRAS geactiveerd?
- Wat is het resultaat van de signaaltransductieroute waar KRAS een rol in speelt?
- Wat is het effect van de mutatie in KRAS?
- Is KRAS een oncogen of een tumorsuppressorgen? Licht je antwoord toe.
- Voor KRAS zijn meerdere experimenteel bepaalde structuren beschikbaar. Er is ook een structuur beschikbaar voor het eiwit met de mutatie. Deze structuur heeft PDB identifier ‘4TQ9’. De structuur van het wildtype eiwit heeft PDB identifier ‘4OBE’. Zoek in beide structuren het aminozuur op dat gemuteerd is en bekijk de verschillen in structuur op die plek. Wat valt je op?