Les 3
Leeruitkomsten
- De student kan PCR primers ontwerpen met Primer-BLAST voor Sanger sequencing.
- De student kan de specificiteit van de ontworpen primers controleren met de UCSC in silico PCR tool.
Voorbereiding
In de vorige les hebben we gezien dat het uitgangsmateriaal voor Sanger sequencing vaak een PCR product is. In deze les leer je hoe je PCR primers kunt ontwerpen.
Biologische achtergrondinformatie
Polymerase chain reaction (PCR)
De PCR (polymerase chain reaction) is een veelgebruikte techniek voor het amplificeren van specifieke stukken DNA. PCR maakt gebruik van primers (een forward en een reverse primer) die het begin en het einde van de te kopiëren DNA sequentie afbakenen. De primers worden door de DNA polymerase gebruikt als startpunt voor de DNA amplificatie. Tijdens de PCR wordt het DNA meerdere keren gekopieerd afhankelijk van het aantal PCR cycli. Elke PCR cyclus bestaat uit drie stappen:
- Tijdens de denaturatie worden de twee DNA strengen van het dubbelstrengs DNA gescheiden van elkaar, zodat enkelstrengs DNA ontstaat.
- Tijdens de annealing binden de primers aan het DNA.
- Tijdens de elongatie wordt het DNA fragment tussen de forward en de reverse primer gekopieerd.
Het resultaat van de PCR is een groot aantal kopieën van het DNA fragment waarin je geïnteresseerd bent.
Ontwerpeisen voor PCR primers
Als je primers ontwerpt voor een PCR dan moeten de primers voldoen aan een aantal voorwaarden:
De primers moeten specifiek binden aan het DNA dat we willen kopiëren en niet op andere plaatsen in het DNA. We willen namelijk zeker weten dat alleen het DNA waarin we geïnteresseerd zijn wordt gekopieerd.
De primers moeten tussen de 18 en 24 basen lang zijn. Als ze korter zouden zijn, wordt de kans groter dat de primers ook op ongewenste plekken in het DNA binden. Als de primers langer zouden zijn, duurt de annealingstap van de PCR erg lang.
De primers moeten een GC clamp hebben. Een GC clamp bestaat uit 1 of 2 G/C nucleotiden aan het 3’ uiteinde van de primer. G en C nucleotiden vormen drie waterstofbruggen, terwijl A en T nucleotiden er maar twee vormen. Door een paar G of C nucleotiden aan het uiteinde van de primer te plaatsen, verbeter je de binding met het DNA.
Het percentage G en C nucleotiden in de primers moet 45-55% zijn. Dat betekent dat ongeveer de helft van de nucleotiden in de primer een G of een C moet zijn.
De meltingtemperatuur van de primers moet groter zijn dan 50 graden Celsius, maar kleiner dan 65 graden Celsius. De meltingtemperatuur is de temperatuur waarbij 50% van de primers gebonden is aan het DNA en 50% niet gebonden is. Tijdens de annealingstap van de PCR moet de temperatuur onde de meltingtemperatuur van beide primers worden gebracht.
De primers moeten geen secundaire structuren vormen of met elkaar hybridiseren.
Er zijn speciale tools die gebruikt kunnen worden voor het ontwerp van PCR primers en die controleren of de primers voldoen aan de bovenstaande ontwerpeisen. In deze les gebruiken we de tool Primer-BLAST.
De genomische sequentie voor een gen opzoeken in NCBI
In les 1 heb je geleerd hoe je de nucleotidensequentie voor transcripten van een gen kunt opzoeken in de NCBI nucleotide database. De sequenties van transcripten bevatten alleen exonen. Als we een PCR willen uitvoeren voor Sanger sequencing, voeren we die PCR uit op genomisch DNA. Genomisch DNA bevat naast exonen ook intronen. Om goede primers te kunnen ontwerpen voor de PCR, moeten we dus werken met de nucleotidensequentie voor het genomisch DNA. Ook deze sequentie is te vinden in de NCBI nucleotide database. We zullen dat hier laten zien aan de hand van een voorbeeld.
Stel dat we willen onderzoeken of een patiënt met taaislijmziekte een mutatie heeft in exon 11 van het CFTR gen. We willen dit bepalen met Sanger sequencing en hebben hiervoor een PCR product nodig. De PCR wordt uitgevoerd op het genomisch DNA van de patiënt en we moeten dus voor het CFTR gen de genomisch DNA sequentie opzoeken. In de onderstaande video wordt getoond hoe je dat doet:
De genomische DNA sequentie voor CFTR heeft in de NCBI nucleotide database de identifier NG_016465.4. In deze sequentie bevindt zich exon 11 op positie 98681-98872.
Opdracht 3
Zoek de coördinaten op voor exon 4 van het CFTR gen.
Klik hier voor het antwoord
Positie 70116-70331 opNG_016465.4.
Opdracht 3
Mutaties in het BRCA2 gen kunnen resulteren in borstkanker. We willen onderzoeken of er mutaties aanwezig zijn in exon 4 van het BRCA2 gen van een patiënt. We willen hier primers voor ontwerpen. Zoek hiervoor de gegevens op voor dit exon.
Klik hier voor het antwoord
Exon 4 van het BRCA2 gen ligt op positie 14597-14705 op NCBI sequentieNG_012772.3.
PCR primers ontwerpen voor een exon
Voor de Sanger sequencing is een PCR product van 400-600 bp nodig. Als we de sequentie van een exon willen bepalen, moet de sequentie van het exon het midden zijn van het PCR product (zie ook het onderstaande figuur). We kunnen dit bereiken door het onderstaande stappenplan uit te voeren:
Bepaal de coördinaten van exon 11 in de genomische sequentie. We hebben de coördinaten al eerder bepaald, namelijk positie 98681-98872 op NCBI sequentie
NG_016465.4.Bepaal het midden van exon 11. We kunnen dit doen door het gemiddelde te bepalen van de start- en eindpositie van het gen (en eventueel af te ronden op een heel getal):
\[ \frac{98681 + 98872}{2} = 98776.5 \approx 98777\]
- Creëer rondom het middelpunt een PCR regio van 600 basen door 300 basen ervoor en 300 basen erna te nemen.
\[ 98777 - 300 = 98477 \\ 98777 + 300 = 99077 \]
Voor exon 11 wordt de PCR regio dus 98477-99077.
- Voor het ontwerpen van de primers definiëren we een zoekgebied van 100 basen aan de uiteinden van de PCR regio. Voor de forward primer wordt dit 98477-98577 en voor de reverse primer 98977-99077.
Als het niet lukt om met Primer-BLAST primers te vinden in de zoekgebieden van 100 basen, dan kun je het zoekgebied uitbreiden van 100 naar 150 of 200 basen.
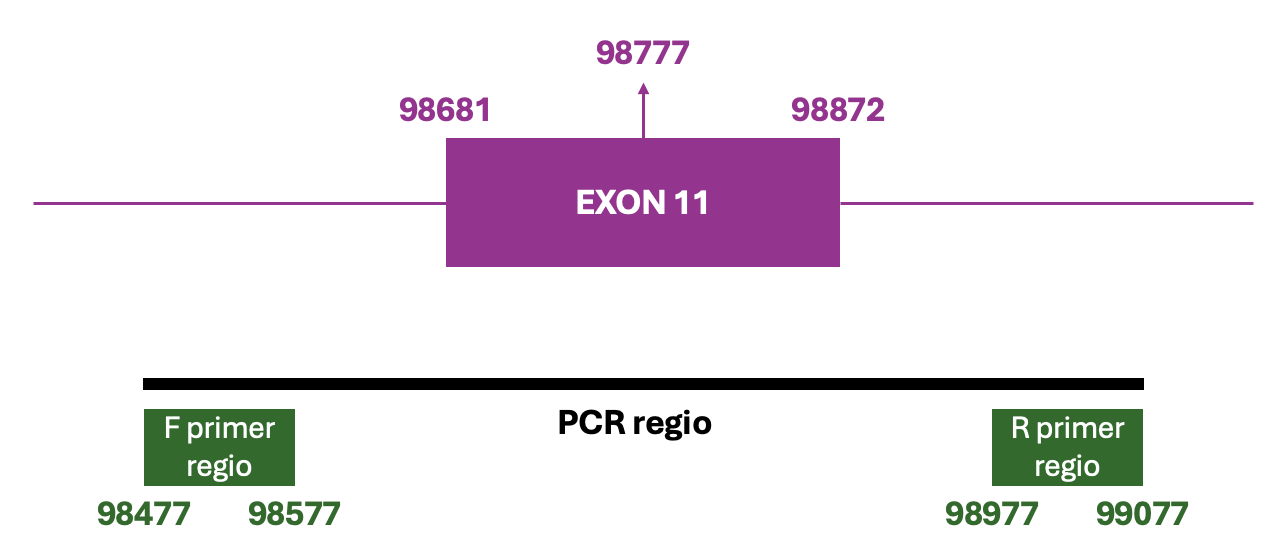
NG_016465.4. Het midden van het exon is op positie 98777. Voor de forward primer definiëren we een zoekgebied van 100 basen tussen positie 98477 en 98577. Voor de reverse primer definiëren we een zoekgebied van 100 basen tussen positie 98977 en 99077.Opdracht 3
We willen PCR primers ontwerpen om met PCR exon 4 van het CFTR gen te kunnen amplificeren. Bepaal de coördinaten voor de zoekgebieden voor de forward en de reverse primer. Gebruik hiervoor de eerder gevonden coördinaten van exon 4.
Klik hier voor het antwoord
Forward primer: 69924-70024 opNG_016465.4.Reverse primer: 70424-70524 op
NG_016465.4.
Opdracht 3
We willen PCR primers ontwerpen om met PCR exon 4 van het BRCA2 gen te kunnen amplificeren. Bepaal de coördinaten voor de zoekgebieden voor de forward en de reverse primer. Gebruik hiervoor de eerder gevonden coördinaten van exon 4.
Klik hier voor het antwoord
Forward primer: 14351-14451 opNG_012772.3.Reverse primer: 14851-14951 op
NG_012772.3.
In de onderstaande video wordt getoond hoe je met Primer-BLAST de primers voor exon 11 kunt ontwerpen:
De output van Primer-BLAST is een lijst met primerparen die gebruikt kunnen worden voor PCR. De output bevat de volgende gegevens:
- De lengte van de primers. De lengte moet tussen de 18 en 24 basen zijn.
- De start- en eindpositie van de primers in opgegeven NCBI sequentie.
- De meltingtemperatuur van de primers. Deze temperatuur moet voor de forward en de reverse primers ongeveer hetzelfde zijn.
- Het percentage G en C nucleotiden in de primers. Dit percentage moet tussen de 45-55% liggen.
- Twee getallen die iets zeggen over de vorming van secundaire structuren (
self complementarityenself 3' complementarity). Deze getallen moeten zo laag mogelijk zijn.
PCR primers controleren met UCSC In Silico PCR
De primers die je hebt gevonden met Primer-BLAST zouden in principe allemaal gebruikt kunnen worden voor het uitvoeren van de PCR. Om meer zekerheid te krijgen over de specificiteit van de primers, kunnen we de UCSC In Silico PCR tool gebruiken. In de onderstaande video wordt getoond hoe je deze tool kunt gebruiken:
Door een in silico PCR uit te voeren met de gevonden primers krijg je een idee over de specificiteit van de primers. Als de in silico PCR maar één product oplevert, kun je de primers in principe gebruiken voor je experiment. Het is wel belangrijk dat je altijd ook op het lab controleert of de PCR maar één product oplevert door met de primers een PCR uit te voeren en het PCR product te analyseren met gel electroforese. Geen enkel computerprogramma kan deze test op het lab vervangen.
Opdracht 3
Ontwerp primers voor exon 4 van het CFTR gen m.b.v. Primer-BLAST. Controleer een primerpaar met de UCSC in silico PCR op specificiteit.
Klik hier voor het antwoord
De Primer-BLAST levert maar een primerpaar op en dit primerpaar is niet ideaal (bijv. omdat een primer langer is dan 24 basen en het percentage GC voor beide primers lager is dan 45%):
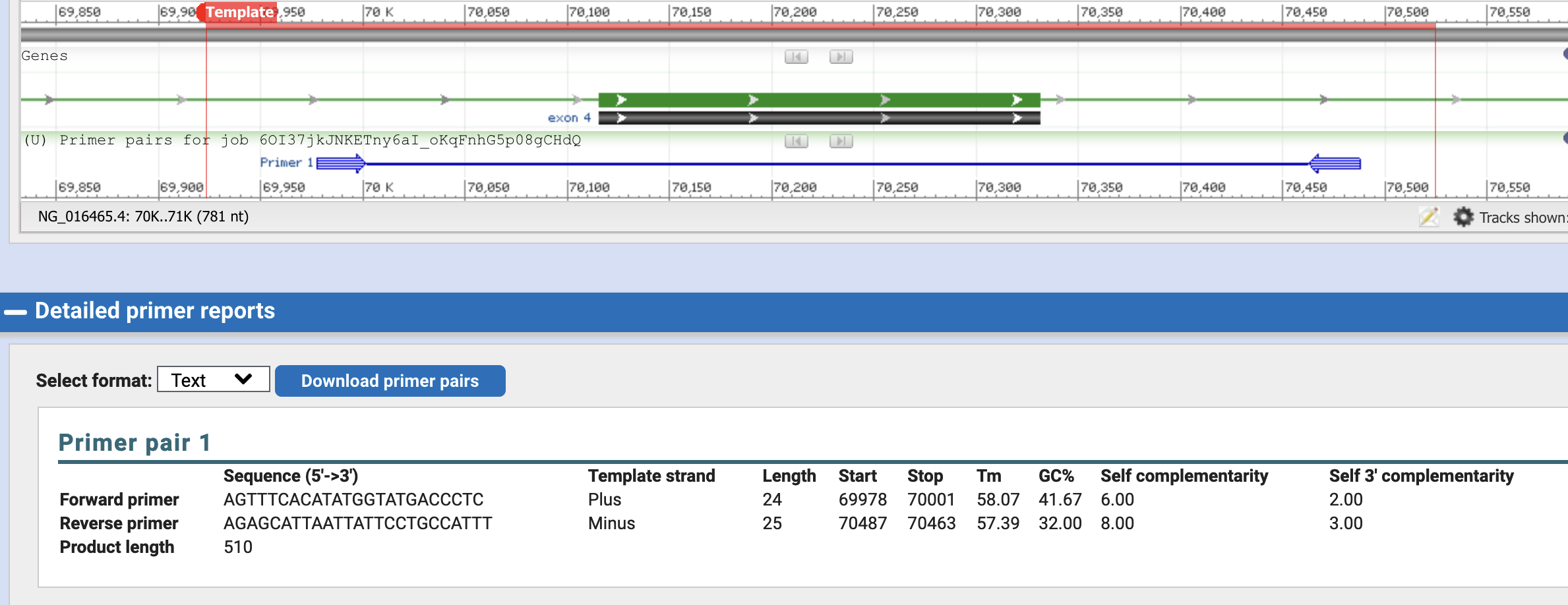
Wel levert dit primerpaar volgens de in silico slechts één product op, wat aangeeft dat de primers specifiek zijn.
In dit geval zouden we het zoekgebied beter kunnen vergroten naar 150 basen. De zoekgebieden voor de primers worden dan als volgt:
- Forward primer: 69924-70074 op
NG_016465.4.
- Reverse primer: 70374-70524 op
NG_016465.4.
Opdracht 3
Ontwerp primers voor exon 4 van het BRCA2 gen m.b.v. Primer-BLAST. Controleer een primerpaar met de UCSC in silico PCR op specificiteit.
Klik hier voor het antwoord
Primer-BLAST vindt meerdere primers. We kiezen een primerpaar die voldoet aan de ontwerpeisen (het eerste primerpaar):

Samenvatting: Mutaties detecteren
In de onderstaande video wordt een overzicht gegeven van de verschillende stappen die nodig zijn om een mutatie te detecteren en annoteren in een patiënt:
Wees je ervan bewust dat we mutaties opsporen in het DNA van de patiënt. Dit betekent dat we een PCR moeten uitvoeren op het DNA van de patiënt en dat we de PCR primers dus moeten ontwerpen voor de DNA sequentie van het gen. We hebben hiervoor dus een NCBI nucleotide ID nodig die begint met NG_.
Als we de DNA sequentie bepaald hebben met Sanger sequencing, willen we het effect van de mutatie op het eiwit bepalen. We hebben hiervoor de CDS van het transcript nodig dat codeert voor de eiwitisoform waarin we geïnteresseerd zijn (vaak is dat de meest voorkomende isoform). Voor deze stap hebben we dus een NCBI nucleotide ID nodig die begint met NM_.
Werkcollege
Casus Les 3
Neuroblastoma is een zeldzame vorm van kanker die bij jonge kinderen voorkomt. Deze vorm van kanker ontstaat in cellen van het zenuwstelsel. Mutaties in het gen ALK kunnen neuroblastoma veroorzaken. Eén van deze mutaties bevindt zich in exon 25.
Voor een patiënt met neuroblastoma willen we bepalen of exon 25 van het ALK gen gemuteerd is. We willen dit doen met Sanger sequencing.
Voer de volgende opdrachten uit voor deze casus:
Zoek algemene informatie op over het ALK gen:
- Op welk chromosoom is ALK aanwezig?
- Hoeveel exonen heeft ALK?
- In welke weefsels komt ALK vooral tot expressie?
- Wat is de functie van het eiwit waar ALK voor codeert?
Ontwerp PCR primers voor exon 25 van het ALK gen. Maak hiervoor gebruik van Primer-BLAST en kies uit de gevonden primers drie primerparen die het beste voldoen aan de ontwerpeisen. Controleer voor deze primers de specificiteit met de in silico PCR tool van UCSC.
De PCR primers worden besteld en alle drie de primerparen blijken in het lab te werken. Eén van de primerparen wordt gebruikt om een PCR uit te voeren op het DNA van de patiënt. Vervolgens wordt er Sanger sequencing uitgevoerd op het PCR product. De resultaten zijn de vinden in het FASTA bestand
casus_les03_sangersequencing_patient.fa. Bepaal of er een mutatie aanwezig is in exon 25 van de patiënt en zo ja, wat het effect van de mutatie is op het eiwit? (Gebruik voor het detecteren van de mutatie transcript 1 van het ALK gen.)